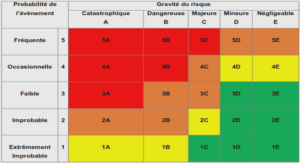
Réaction de polymérisation en chaine (PCR)
Dans cette section nous pressentons la technique d’amplification d’une séquence d’ADN qui la PCR.
Avant d’expliquer les différentes techniques de séquençage nous explicitons d’abord la PCR.
La réaction de polymérisation en chaine (PCR) est une technique qui amplifie la séquence d’ADN (réplication). Cela permet de dupliquer à plusieurs millions d’exemplaires un fragment d’ADN grâce à l’ADN polymérase. Cette technique utilise des cycles de trois phases au cours desquelles chaque double brin et dénaturé et séparé de sa polymérase (qui est maintenu par des protéines). Une variation spécifique de la température aux nombreuses amorces présentes en suspension dans la solution de l’hybrider aux simples brins. Sans que ceux- ci ne s’hybrident entre eux (ce qui rendait impossible l’accrochage de l’amorce).
Enfin la polymérase assimile progressivement les nucléotides néoformés libres ajoutés également dans la solution jusqu’à obtenir de double brins complets, c’est l’élongation. Le cycle Cryptographie avec les ADN: cas des systèmes symétrique Année 2020 d’une durée maximum de quatre minutes, et répète en boucles le nombre de brins à chaque fois (suite logique de puissance 2). La dénaturalisation (ou déshydratation), c’est-à-dire la rédaction de deux brins composant la molécule d’ADN, est obtenu par deux brins composant la molécule d’ADN, est obtenu par élévation de température ce qui a pour effet de fragiliser les liaisons hydrogènes reliant les bases azoté.
Méthode d’automatisation du séquençage.
Les deux premières techniques de séquençage ont été développées en parallèle au milieu des années 1970 par Sanger (Grande-Bretagne) et Gilbert(Etats-Unis) ont toutes deux été récompensées d’un prix Nobel de chimie en 1980. Nous avons en premier lieu la méthode de Sanger, qui consiste à introduire en parallèle des brins d’ADN cibles dénaturés en grand nombre des amorces (sur laquelle se fixe la polymérase) dans quatre tubes à essais distincts. Les ADN polymérases sont capables de synthétiser un brin complémentaire d’ADN, à partir d’un brin matrice. Pour le séquençage des nucléotides légèrement différents sont utilisés : les diesoxyribonucleotides (ddNTP) au lieu des desoxyribonucleotides triphosphate (dNTP). Les ddNTP diffèrent des dNTP par l’absence d’un regroupementOH en position 3’. Ainsi lorsqu’une ADN polymérase utilise un ddNTP, elle n’est plus capable de rajouter le moindre nucléotide à sa suite. La synthèse du brin d’ADN s’arrête. En général les protocoles utilisé pour séquencer un fragment d’ADN, il faut préparer 4 mélanges :
I Le fragment qui doit être séquencé ;
I Un petit morceau d’ADN dont la séquence est complémentaire à l’extrémité 3’ du fragment à séquencer ;
I Les 4 dNTP’s (dCTP, dATP, dGTP et dTTP) ;
I L’ADN polymérase ;
I Dans chaque tube, des petites quantités d’un ddNTP fluorescent ou radioactif.
L’incorporation aléatoire d’un ddNTP stoppe la synthèse. On obtient à la fin des réactions un ensemble des brins d’ADN de tailles variées selon l’endroit ou ddNTP se sera inséré et que la réaction aura ainsi été stoppée. L’utilisation de d’un ddNTP permet d’obtenir un ensemble de fragment d’ADN de taille différentes correspondant aux emplacements d’un nucléotide donnée.
Par contre à celle de Gilbert est une réaction chimique qui ne modifie aucune base elle permet de couper le brin suite à un type de base. Étant donnée le nombre important de copiés et présentés dans le tube, on obtient statistiquement chaque possibilité. La méthode de l’électrophorèse suit ensuite celle de Sanger. Moins facile à robotiser que la méthode de Sanger, cette technique est aujourd’hui’hui très peu utilisée dans le milieu industriel. Aujourd’hui’hui, la plus part des séquençages sont réalisés par des séquenceurs industriels entièrement automatisé. Ceux-ci utilisent la technique de Sanger mais avec des méthodes de révélation différente. Les fragments d’ADN sont masqués par des marqueurs fluorescents : Leur taille est en suite déterminée par chromatographie ou électrophorèse assistée par Ordinateur.Avec ce techniques, On peut séquencer jusqu’à 1000 bases avec les meilleurs séquenceurs contre 200 à 300 via une méthode manuelle comme celle exposées ci-dessus.
Alignement des séquences
Dans cette section nous élaborons l’alignement des séquence d’ADN .
La biologie moléculaire est une discipline scientifique qui vise à comprendre les mécanismes de fonctionnement de la cellule au niveau moléculaire. En bio-informatique, l’alignement de séquences ou alignement séquentiel est une manière de représenter deux ou plusieurs séquences de macromoléculaire biologiques (ADN, ARN ou protéines) les unes sous les autres, de manière à en faire ressortir les régions homologues ou similaires. L’opération d’alignement sert à identifier des zones communes à un groupe k séquences.
Conclusion
Nous avons abordé dans ce chapitre beaucoup des notions plus particulièrement la réaction en chaine polymerase PCR, Méthodes de séquençage de l’ADN ainsi que le codage d’ADN en ADN numérique.
Le chapitre suivant portera sur l’etat de l’art de la cryptographie d’ADN.