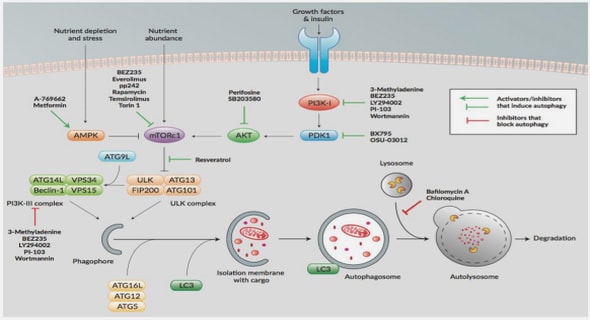
Reconnaissance de motifs dans les séquences biologiques
Dans ce chapitre, nous présentons les notions utilisées dans cette thèse. Tout d’abord, nous rappellerons le vocabulaire lié à la reconnaissance de motifs. Ensuite, nous détaillerons plusieurs éléments de base liés à la biologie et ce que cela peut impliquer concernant la modélisation. Nous passerons en revue différents outils de reconnaissance de motifs communément employés en biologie, qui illustrent différentes grandes approches en la matière.
Nous enchaînerons ensuite sur les modèles grammaticaux, en présentant tout d’abord différents principes qui y sont liés : les notions de grammaire et de langage, les différents niveaux de grammaires et l’expressivité qui en découlent et enfin l’évocation de la façon dont les grammaires peuvent intervenir dans la recherche de motifs biologiques,
notamment grâce aux grammaires dédiées que sont les grammaires à variables de chaînes (notées SVG, pour String Variable Grammar). Enfin, nous terminerons par une présentation du langage Logol, un langage grammatical à haut niveau d’expressivité, basé sur les SVG, qui constitue le point de départ et le fil rouge de la thèse.
Vocabulaire lié à la reconnaissance de motif Pour commencer, voici quelques définitions du vocabulaire de base utilisé en reconnaissance des motifs [Wol06, EZ11] en biologie. Par ailleurs, plusieurs mots liés à la reconnaissance de motifs peuvent avoir des significations différentes selon les communautés (tel que motif et modèle).
Nous donnons ici la signification que nous avons choisie d’associer à ces mots dans la suite de ce manuscrit. Modèles et motifs Tout d’abord, les termes motifs et modèles, au cœur de cette thèse, prêtent souvent à confusion selon les communautés et le contexte. Dans ce manuscrit, nous utiliserons le mot motif pour désigner la réalité biologique d’une notion ou d’un signal qu’on cherche à identifier sur une séquence.
Il peut s’agir d’un signal simple, tel que, par exemple, un site de fixation, qui est un petit segment d’un brin d’ADN où se fixe une protéine; mais il peut s’agir également d’un signal complexe, tel que, par exemple, le signal de « frameshift-1 » (cf page 22), qui indique qu’un ARN est susceptible d’être traduit en deux protéines différentes,
suite au glissement de la machinerie de traduction. Nous utiliserons le mot modèle pour désigner la représentation concrète d’un motif dans un langage formel (sous la forme d’une expression régulière, d’une matrice, d’une grammaire Logol…).
Ainsi, le modèle d’un site de fixation peut être décrit comme un ensemble de mots possibles, alors que le modèle d’un motif « frameshift-1 » peut être décrit comme la présence sur la séquence de plusieurs signaux successifs (une fenêtre glissante suivie d’une structure en pseudo-noeud etc.). — Alphabet : un alphabet est un ensemble fini de symboles.
Par exemple, L={A, C, G, T} est l’un des alphabets des séquences d’ADN; — Mot:unmot défini sur un alphabet est une séquence finie d’éléments de cet alphabet. Par exemple, « AACGA » est un mot défini sur l’alphabet L; — Mot vide : un mot peut ne contenir aucun caractère. Ce mot vide sera représenté ici par le symbole ϵ; — Référence : une instance déjà connue d’un motif, souvent prouvée biologiquement, utilisée pour définir les spécificités d’un modèle.
Par exemple, les séquences dont on sait déjà que la protéine P peut s’y fixer constituent les références du motif du site de fixation P; — Modèle : un ensemble de règles et de contraintes permettant de définir les cibles recherchées lors d’une recherche par reconnaissance de motifs. Par exemple, si les références du motif de fixation de la protéine P vérifient une taille de 10, commencent par A et finissent par T, le modèle du site de fixation pourra être le cumul de ces trois règles;
Reconnaissance de motifs ou Pattern matching : Reconnaissance exacte de motifs : pour un modèle M et un texte T, localisation de toutes les occurrences de M dans T. Reconnaissance approchée de motifs approchée : pour un modèle M, un texte T, une fonction de similarité basée sur une distance d et un paramètre seuil k, localisation des segments I du texte T tels que d(M,I) ≤ k. — Background : proportion d’apparition des différentes lettres dans un texte. Les back grounds les plus détaillés donne la proportion d’apparition d’une lettre en fonction des k lettres précédentes (Chaînes de Markov d’ordre k [PP97]).
Le background permet de juger de la rareté d’un mot par rapport au hasard en prenant en compte d’éventuels biais de composition des séquences. Dans un background riche en A, la localisation d’un mo tif AAATAA est moins significative que dans un background avec une équi-répartition des lettres; — Match ou Hit : une instance dans un texte identifiée par un modèle. Par exemple : ATGCGCACGT est une instance reconnue par le modèle du site de f ixation de la protéine P : elle commence par A, fini par T et fait une taille de 10;
— Insertion : Ajout d’un élément dans une instance par rapport à une référence. Par exemple : ATGCATG contient une insertion en position 4 par rapport à la référence ATGATG — Délétion : Suppression d’un élément dans une instance par rapport à une référence. Par exemple : ATG_TG contient une délétion en position 4 par rapport à la référence ATGATG — Substitution :
Remplacement d’un élément par un autre dans une instance par rapport à une référence. Par exemple : ATGTTGcontient une substitution en position 4 par rapport à la référence ATGATG — Gap : Symbole d’une insertion ou d’une délétion entre une séquence et une référence, notamment lors d’un alignement.