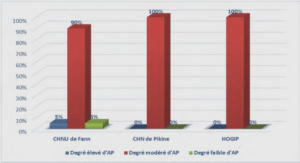
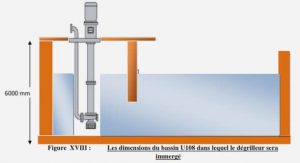
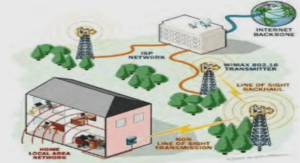
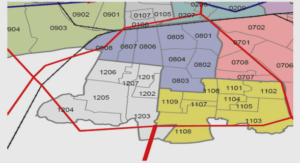
Modèles et modélisation multi-niveau
Modélisation multi-niveau
La modélisation multi-niveau est un terme que nous avons adopté pour rendre compte des techniques de modélisation couplant plusieurs modèles pour décrire un système. Dans ce contexte, un modèle propose d’observer le système étudié à travers le prisme d’un point de vue particulier, appelé niveau de description. La notion de niveau met ici l’accent sur le fait que chaque modèle peut ne s’intéresser qu’à une sous-partie du système (peu importe ce que l’on entend par sous-partie ici) et propose donc une description incomplète du système.
C’est par le couplage entre les modèles que ces descriptions complémentaires se complètent mutuellement, pour finalement obtenir une description plus pertinente du système dans son ensemble. Ainsi, la modélisation multi-niveau met en avant une méthodologie générique pour l’élaboration d’un modèle par composition (d’autres modèles). Dans cette section, nous nous intéressons à différents exemples de composition de modèles pour en analyser les difficultés. Nous en concluons que la notion d’identité est au cœur de la modélisation multi-niveau.
Couplage de modèle
Dans cette section nous nous intéressons à deux exemples types de couplages de modèles. Le premier exemple concerne la modélisation intégrative du phénotype d’une cellule biologique entière à partir des modélisations des différents mécanismes moléculaires qui la composent. Le second exemple s’intéresse à la morphogénèse d’une communauté de villes à partir de modélisations orientées quartier. Dans ces deux exemples, des modèles élémentaires sont utilisés pour en conjuguer les points de vue et obtenir une description plus complète du système. Dans les deux cas également, un changement d’échelle s’opère, entre les considérations micro(scopiques) des modèles élémentaires, et le niveau macro(scopique) du système étudié.
En modélisation, l’approche intégrative consiste à construire des modèles en cherchant à expliquer simultanément plusieurs phénomènes, permettant de révéler des dynamiques n’apparaissant pas dans des approches indépendantes. Ces modèles sont construits par l’accumulation de modèles préexistants jugés valides et par l’établissement d’un réseau de communication entre ces modèles, voire la conception d’adaptateurs permettant de « traduire » la sortie d’un modèle en l’entrée d’un autre. Cette approche est particulièrement développée dans le monde de la recherche sur le vivant [18], et permet des échanges prolifiques entre les nombreuses disciplines de la biologie (généticiens, physiologistes, comportementalistes, paléontologistes, écologistes, etc.)
et donc entre les nombreuses perspectives d’un même système qu’il est possible de récolter. De nombreux projets utilisent cette approche pour comprendre le fonctionnement d’un organisme biologique complet, comme par exemple une cellule. À titre d’exemple, un modèle computationel d’une cellule entière de Mycoplasma genitalium est proposé dans [21]. Ce modèle donne l’expression complète du phénotype de la cellule à partir de son génome en formulant des hypothèses sur de nouveaux mécanismes biologiques. L’approche de ce projet passe par le concept de modularité biologique qui permet de déterminer une liste d’observables du système dont les valeurs correspondent à des états de la cellule.
Plusieurs modèles permettent d’expliquer séparément comment certaines observables sont liées entre elles. Une fois ces modules établis, les modèles sont assemblés en suivant les relations entre les observables, puis ajustés pour respecter les contraintes physiques et biologiques du système dans son ensemble. Les solutions élaborées durant ce type de projet sont pour le moment ad hoc et la recherche de méthodes génériques pour rendre cette approche utilisable pour la description d’autres systèmes biologiques est en cours. Le projet précédent a par exemple mis en lumière, dans une seconde publication [27], une liste de sept limites à dépasser pour généraliser leur approche intégrative à d’autres organismes.
Parmi ces points, les suivants, attireront plus particulièrement notre attention car ils évoquent les limites des outils pour la modélisation intégrative : Agrégation des données L’acquisition des données disponibles dans la littérature a été compliquée par l’absence d’une source unifiée permettant de compiler l’ensemble des connaissances sur un sujet en particulier, dans ce cas sur l’organisme étudié. La modélisation d’une cellule entière bénéficierait grandement d’outils permettant d’agréger et de normaliser toutes les données nécessaires à la calibration des modèles ; Construction et intégration de modèles Un facteur limitant a été la disparité des types de modèles décrivant à différentes échelles d’espace et de temps l’organisme.
Unifier la modélisation intégrative passerait donc par une généralisation du concept de modèle à travers un cadre mathématique commun à l’ensemble de la modélisation. Comprendre et analyser les relations entre modèles dépend de l’existence ce cadre commun ; Calcul accéléré La simulation numérique est un outil clef de la réussite de ce projet. Chaque simulation de Mycoplasma genitalium a été très consommatrice en temps de calcul, ce qui est un facteur limitant pour la réussite des projets futurs.
Élaborer des modèles d’organismes plus complexes dépendra donc fortement de la capacité des simulateurs à explorer une large portion de l’espace des paramètres dans un temps plus restreint. Le calcul général sur carte graphique, via un framework tel que CUDA ou OpenCL, est une piste à explorer pour améliorer l’efficacité des simulateurs