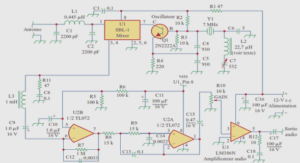
Application à l’étude de l’état redox des cystéines
Les résultats de la comparaison de la méthode FASP à la précipitation acide (TCA) (chapitre 5) pour l’application du protocole OcSILAC ont montré que la méthode FASP n’induisait pas d’avantage d’oxydation durant le protocole de traitement. Ces observations ont motivé le choix de concevoir un dispositif microfluidique destiné à la miniaturisation du protocole OcSILAC. Le ChipFilter a donc été conçu, testé et utilisé pour le traitement d’une protéine standard et ensuite d’un échantillon complexe. Une sensibilité satisfaisante a été constatée avec l’analyse de la BSA. Ensuite, l’analyse d’extrait protéique de cellules de levure a montré une bonne couverture du protéome. L’échantillon d’extrait protéique de levure a ensuite été utilisé pour tester la miniaturisation du traitement de l’échantillon dans le ChipFilter.
Dans ce chapitre sont présentés les résultats de la comparaison du protocole OcSILAC effectué dans le ChipFilter par rapport à la méthode classique caractérisée par la précipitation acide (TCA) et dont la miniaturisation était l’objectif principal de cette thèse. Malgré la sensibilité du ChipFilter, une quantité d’échantillon de 100 µg a été choisie. Ce choix est motivé par le besoin de se mettre un peu au niveau de la sensibilité de la méthode classique afin de pouvoir comparer la couverture du protéome et une éventuelle oxydation artéfactuelle. Ce chapitre comprend aussi la description du couplage du ChipFilter pour l’amélioration de la sensibilité et de la reproductibilité.
Miniaturisation du traitement de l’échantillon pour l’étude redox
L’étude des PTMs de la cystéine est une analyse complexe du fait de la grande réactivité des thiols et du caractère labile de ces modifications qui peuvent être affectées durant la préparation de l’échantillon. L’acide trichloroacétique TCA) est en général utilisé pour passer en conditions acides et bloquer la réactivité des thiols durant l’extraction des protéines ou l’élimination d’un réactif en excès. Mais la protéolyse pour générer les peptides en bottom-up nécessite de neutraliser l’échantillon. L’élimination du TCA passe d’abord par une précipitation des protéines suivi d’un rinçage avec de l’acétone froid. Ensuite l’évaporation de l’acétone permet de reprendre les protéines avec un tampon approprié permettant le blocage définitif des thiols. C’est une approche très utilisée malgré les risques de modification artéfactuelle. L’utilisation du ChipFilter pour des applications en rédoxomique est intéressante dans le sens où le rinçage de réactifs en excès peut se faire rapidement dans des conditions réduisant le risque de modifications artéfactuelles.
La dernière étape du protocole OcSILAC ne peut être réalisée sur le ChipFilter car ce dernier n’est pas équipé pour la rétention spécifique des peptides marquées avec la biotine. Après l’étape d’élution, les peptides récoltés vont faire l’objet d’un fractionnement sur une colonne de streptavidine dans les mêmes conditions que dans le protocole OcSILAC classique. Pour comparer la préparation d’échantillon dans le ChipFilter et le protocole OcSILAC classique, 100 µg d’extrait protéique de cellule de levure ont été traités par ChipFilter et autant par le protocole OcSILAC classique caractérisé par la précipitation acide. D’un côté, toutes les étapes de préparation des protéines et leur digestion ont été effectuées sur puce.
La précipitation acide des protéines n’est plus nécessaire pour éliminer les réactifs en excès car ces molécules passent facilement à travers la membrane. Pour le protocole OcSILAC classique, les protéines sont précipitées à la fin de chaque réaction pour éliminer les réactifs en excès. Les peptides générés sont passés sur une colonne de streptavidine dans le but d’enrichir sélectivement les peptides portant une cystéine biotinylée. Dans les 2 cas, cet enrichissement est effectué dans les mêmes conditions. Les fractions enrichies(contenant les peptides initialement oxydés et biotinylés) et non enrichies (les autres peptides) ont été analysées séparément. Une quantification sans marquage (LabelFree) a ensuite été effectuée séparément dans les fractions enrichie et non enrichie pour comparer le nombre de peptides identifiés ainsi que leur intensité.
Identification et quantification
Pour résumer, la même quantité d’échantillon a été traitée dans le ChipFilter et par leOcSILAC sauf le fractionnement qui est effectué dans les mêmes conditions que le protocole classique. Les fractions issues de chaque approche sont analysées séparément. Nous allons donc avoir, pour chacune des 2 approches que nous comparons, une fraction enrichie ou Bound et une fraction non enrichie ou UnBound. La fraction enrichie contient principalement les peptides contenant une cystéine marquée par la biotine clivable et soluble dans l’eau (donc cystéine oxydée). Ces peptides ont été retenues spécifiquement sur la colonne de streptavidine d’où le nom (Bound, lié). La fraction non enrichie correspond à la partie de l’échantillon qui n’a pas été retenue par la colonne de streptavidine et qui contient donc tous les autres peptides. Les résultats de cette comparaison basée sur les peptides et protéines identifiés dans ces fractions ainsi que leurs intensités sont présentés dans cette partie.
Le premier point de comparaison concerne la couverture du protéome avec le nombre de protéines et de peptides identifiées. Le tableau 10 montre le nombre de peptides et de protéines identifiés dans les fractions enrichies (Bound) et non enrichie (Unbound). On observe que dans la fraction enrichie, le nombre de protéines et de peptides identifiés est plus élevé pour l’approche classique (TCA_en_solution) que pour la digestion sur puce (ChipFilter). C’est le contraire qui est observé dans la faction non enrichie.