Biopuces a ADN par fluorescence
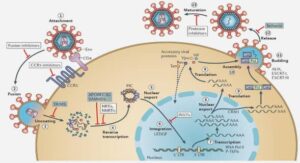
Les micro-arrays [18], ou puces a ADN, font partie des biopuces les plus connues et les plus utilisees. Elles permettent d’analyser l’expression de milliers de genes simultanement et a tres haut debit. Ce type de systeme est utilise pour le diagnostic génétique ou l’analyse du transcriptome. Le principe de fonctionnement des puces a ADN est relativement simple. Il est base sur les mecanismes de transcriptase inverse des ARN (souvent appele Reverse-Transcriptase ou RT en anglais). Une cellule vivante contient en permanence un ensemble de molecules d’ARN de differents types, dont les ARN messagers (ARNm). Ces ARNm sont le produit de la transcription dans le noyau de fragments d’ADN presents dans les genes. Apres maturation (permettant d’eliminer les introns par epissage), les ARNm « matures » migrent dans le cytoplasme ou il sont traduits par les ribosomes, et donnent des proteines. L’idee est donc d’extraire les ARNm des cellules a etudier, puis d’effectuer une transcription inverse (RT), grˆace a une enzyme specialisee, en mˆeme temps qu’une amplification par reaction en chaˆıne de polymerase (Polymerase chain reaction, ou PCR). La methode de transcription-amplification est donc nommee RT-PCR.
Le produit final contient donc des fragments d’ADN complementaires (nommes ADNc) au fragment de gene exprime. La quantite de ADNc presente est donc directement proportionnelle a la quantite d’ARNm transcrite, et il devient donc possible de remonter au niveau d’expression des genes de la cellule. Neanmoins, les ADNc ne nous renseignent pas par eux mˆemes sur le niveau d’activite d’un gene ou d’un ensemble de genes en particulier. Pour cela, il est necessaire de marquer les fragments d’interˆet et de les « retenir » afin d’effectuer un tri par rapport aux multiples fragments existants. L’aspect novateur des puces a ADN consiste donc en la methode de caracterisation de ces niveaux d’expression. Les ADNc obtenus (les molecules cibles) vont ˆetre marques avec un fluorophore, par exemple Cy3 pour le contrˆole et Cy5 pour l’echantillon a analyser, puis melanges ensemble en quantites egales. La solution sera ensuite mise en contact avec une puce a ADN.
Sur la puce sont deposes un ensemble de zones d’interaction, constituees de fragments d’ADN codants pour diverses proteines d’interˆet par exemple. Ces fragments d’ADN sondes sont disposes sur une lamelle de verre fonctionnalisee chimiquement. C’est l’interaction sonde-cible qui va permettre de cartographier le niveau d’expression d’un gene ou d’une proteine. En effet, les ADNc obtenus vont parfaitement s’hybrider avec le fragment de gene depose, alors qu’ils vont moins fortement s’hybrider aux autres fragments sondes. Apres rin¸cage, seuls les brins parfaitement complementaires resteront sur la surface de la puce. Il est enfin possible de « lire » la puce en utilisant 2 lasers et un capteur CCD : un laser emettant dans le vert (a 540 nm) qui relevera le niveau de fluorescence des ADNc contrˆole, et un laser emettant dans le rouge (a 650nm) qui relevera le niveau de fluorescence des ADNc de l’echantillon. En utilisant des algorithmes de recalage d’images, il est possible de superposer les 2 images, et ainsi obtenir une image a 2 couleurs, contenant des informations sur le niveau d’expression relatif du contrˆole et de l’echantillon. Les syst`emes de pucesa ADN par fluorescence ont connus un developpement tr`es important depuis les premi`eres membranes nylon heritees du Southern blot [19] dans les annees 80 [20].
Tout d’abord, les membranes souples ont ete delaissees pour des plaques de verre, permettant l’observation de la fluorescencea travers la puce. L’augmentation de la densite de zones d’interaction par unite de surface induit par le passagea un support solide a permis de passer de quelques dizaines de spots par cm2 dans les annees 1980,a plusieurs centaines de milliers de spots par cm2 grˆacea la synth`ese in-situ de sondes [21, 22] (Affymetrix [23]), ou l’impressiona jet d’encre [24] (Agilent [25] par exemple). Cette augmentation de densite s’accompagne inevitablement d’un accroissement exponentiel du volume de donneesa traiter et stocker. L’analyse d’un tel volume de donnees a donne lieua l a mi se au poi nt de nouveaux al gori t hmes [ 26] eta l’utilisation de techniques issues de la bioinformatique [27]. Un des principaux freinsa l’essor des methodes basees sur les biopucesa ADN par fluorescence est son coˆut. Le coˆut de fabrication de la puce elle-mˆeme (environ 200e pour pour l’achat d’une puce Affymetrix couvrant le genome humain), qui peut ˆetre utilisee pour mesurer les niveaux d’expression de plusieurs echantillons en parall`ele, n’est pas le probl`eme principal. Le coˆut de marquage est sans doute le plus important (de l’ordre de 200e), suivi du coˆut d’hybridation et de traitement des donnees (environ 150e) [28, 29]. De ce fait, il existe une forte reticencea systematiser l’utilisation de biopucesa fluorescence dans les hˆopitaux, et celles-ci restent bien souvent cantonnees aux applications en recherche. Bien que certains syst`emes soient capables de travailler en temps-reel [30] (celui de Genewave [31] par exemple), la lecture des biopucesa fluorescence est dans la majorite des cas limitee par l’aspect statique de la mesure en point-final. En effet, il est necessaire de faire interagir les molecules, puis de rincer la puce pour eliminer l’exc`es de cibles, avant de pouvoir la lire.
Historique des biocapteursa SPR
En 1902, Wood observe de brusques changements d’intensite dans certaines parties du spectre d’une source de lumi`ere de profil continu, reflechi par un reseau metallique [33], se sont les fameuses « anomalies de Wood ». Un aspect frappant des experiences de Wood, est que ces changements ne se produisent que lorsque la lumi`ere poss`ede une polarisation transverse magnetique (TM ou polarisation p, c’esta dire que le champ electrique oscille dans le plan de la figure 5 du paragraphe 1.2.2). C’est plus tard, en 1941, que Fano [34] apporte un fondement theoriquea ces experiences, en demontrant que les anomalies sont duesa des ondes quasi–stationnaires « roulant » le long du metal. En 1958, R. Ferrell [35] decrit theoriquement le phenom`ene de couplage entre une onde electromagnetique et les modes de surface entre un metal et un dielectrique. Ce n’est qu’en 1968 que A. Otto [36] propose une demonstration experimentale du phenom`ene base sur la reflexion totale attenu ee (ATR ou Attenuated Total Reflection). Il utilise un prisme de dielectrique de haut indice pour augmenter le momentum de l’onde incidente, la reflexion ATR sur la base du prisme cree alors une onde evanescente, qui excitea son tour la surface de metal, placee `a une f ai bl e di st ance de l a base du pri sme. La meˆme annee, Kretschmann et Raether [ 37] proposent une configuration similaire, mais o`u le metal est directement depose sur la base du prisme. C’est cette configuration qui sera et reste la plus utilisee dans toutes les applications de la resonance de plasmon de surface de part sa simplicite de mise en place notamment. Pendant une vingtaine d’annees, la resonance de plasmon de surface restera une « curiosite » physique interessante sur le plan theorique, mais peu utilisee dans la pratique.
Figure 4 – Nombre de publications par annee (entre 1982 et 2008) dont les mots cles contiennent « surface plasmon resonance » ou « plasmonic(s) ». (Bleu) Resultats pour l’interrogation de la base ISI Web of Knowledge (incluant les proceedings de conferences). (Vert) Resultats pour la base Medline, plus orientee biomedical et donc plus en aval dans les applications. L’idee d’utiliser la resonance de plasmon de surface en tant que biocapteur ne sera developpee qu’`a partir de 1983, quand B. Liedberg, C. Nylander et I. Lundstrom [38] developpent un syst`eme base sur la configuration de Kretschmann pour detecter de faibles concentrations (quelques ppm) de gaz anesthesique. Ils realisent egalement une experience o`u des γ-globulines adsorbees sur la surface de metal par le biais d’une chimie de surface specialisee sont mises en presence avec des anticorps anti γ-globulines. L’augmentation d’epaisseur et le changement d’indice de refractiona la surface resultant de la liaison antig`ene-anticorps donne lieua un decalage de la courbe angulaire de resonance. Cet article donne le coup d’envoi de l’utilisation du phenom`ene de SPR pour le developpement de biocapteurs [39]. A partir du debut des annees 1990, o`u les premiers syst`emes commerciaux sont fabriqu es (premier syst`eme Biacore en 1990 par exemple), le nombre de publications du domaine explose. La figure 4 illustre ce phenom`ene : d’une cinquantaine en 1990,a plus de 2800 publications en 2008 ! Toutes les recherches ne sont neanmoins pas orientees vers le developpement de biocapteurs, et les applications de la resonance de plasmon de surface sont nombreuses : l’imagerie en dessous de la limite de diffraction [40], le developpement de cristaux plasmoniques [41] pour le guidage adiabatique des ondes de surface, le developpement de nouvelles techniques de lithographie optique [42], la fabrication de « nano-circuits » [43] integres, bases non pas sur l’electronique, mais sur la plasmonique, permettanta terme de reduire de plus d’un ordre de grandeur les tailles des circuits int egres actuels, etc. Dans le reste du chapitre, nous nous concentrerons sur les aspects orientes « biopuces » de la resonance de plasmon de surface, et nous delaisserons toutes les autres applications precedemment citees.
Table des figures |