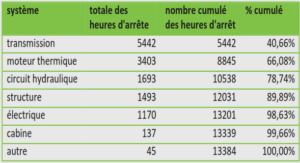
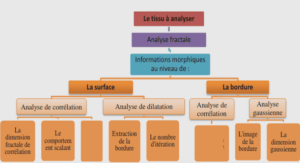
Les « Omics »…
Les Omics recouvrent différents champs d’investigation et nouvelles technologies dans plusieurs domaines : génomique, protéomique, métabolomique… Dans le cadre de ce travail de thèse, nous ne développerons dans cette partie que les Omics se rapportant au domaine des polypeptides et à la thématique des protéases.
Protéomique
Le concept de l’analyse protéomique a été introduit en 1998 par Anderson et Anderson, qui le définissent comme l’analyse quantitative des niveaux d’expression des protéines, visant à déchiffrer les mécanismes du contrôle de l’expression génique [1]. Les études protéomiques peuvent être envisagées à des échelles variées. Avec l’achèvement du Projet de Génome Humain en 2003, la recherche scientifique sur la détermination des protéines du corps humain s’est accentuée. Cette étude de toutes les protéines produites par les cellules et l’organisme implique l’identification des protéines de l’organisme et la détermination de leurs rôles dans les fonctions physiologiques et pathologiques. Le séquençage de nombreux génomes entiers de procaryotes et d’eucaryotes a suscité un regain d’intérêt pour la protéomique.
Le terme protéome désigne l’ensemble des protéines exprimées par un génome donné dans un environnement donné, à un moment donné. Tandis qu’un génome reste globalement inchangé, chaque cellule exprime des protéines particulières de façon radicalement différente en réponse à son environnement. Dans la mesure où ce sont les protéines qui sont directement impliquées dans la plupart des processus biochimiques à la fois normaux et pathologiques, une compréhension plus complète des maladies peut être obtenue en étudiant directement les protéines d’une cellule ou d’un tissu malade. Le protéome est par nature dynamique, ce qui a conduit certains chercheurs à utiliser le terme de protéome fonctionnel pour décrire l’ensemble des protéines exprimées par une cellule spécifique à un temps donné. Alors que le génome humain contient environ 22 000 gènes codant potentiellement pour 40 000 protéines différentes, l’épissage alternatif de l’ARN et les modifications post-traductionnelles augmentent le nombre de protéines ou de fragments de protéines jusqu’à 2 000 000 [3]. En conséquence, le protéome est beaucoup plus complexe que le génome.
Peptidomique
Définition La protéomique s’est établie comme une méthodologie remarquable pour fournir des données sur l’identité des protéines et de leurs fonctions. Cependant, les approches associées sont, pour des raisons méthodologiques, limitées à l’analyse de protéines de masses moléculaires supérieures à 10kDa. Le développement de méthodologies couvrant les peptides et les protéines de plus bas poids moléculaire (0,5 à 15kDa) était nécessaire. En effet, parmi ces peptides, se trouve la famille des hormones, des cytokines et des facteurs de croissance, qui jouent un rôle central dans de nombreux processus biologiques. Pour récapituler les technologies utilisées pour identifier ces peptides, le terme 13 peptidomique (en anglais peptidomics) a été introduit. Cette notion a été utilisée pour la première fois en 2001 par Schulz-Knappe, et définie comme un sous-ensemble de la protéomique pour la description des peptides, considérés, eux aussi, comme produits de gènes [5, 6]. La peptidomique a pour but d’identifier tous les peptides endogènes d’une cellule, d’un organe ou d’un organisme (i.e. le peptidome). La peptidomique se définit comme une méthodologie permettant la description la plus complète possible des peptides d’un échantillon biologique, tant d’un point de vue qualitatif que quantitatif.
Les difficultés de l’analyse : différences avec la protéomique
Bien que la peptidomique et la protéomique partagent de nombreux objectifs scientifiques et de nombreux outils techniques, il y a quelques différences majeures entre ces approches. La plupart des études protéomiques utilisent des enzymes de digestion comme la trypsine pour générer des fragments qui peuvent être séquencés par MS/MS afin d’identifier les protéines présentes dans l’échantillon [7]. A l’inverse, la plupart des études en peptidomique n’utilisent aucune étape de digestion in vitro, afin d’identifier la forme native des peptides, en incluant les modifications post-traductionnelles [5, 8]. Bien que la suppression de cette étape de digestion simplifie la préparation des échantillons, l’analyse des données est généralement plus délicate pour les gros peptides. En protéomique, il n’est pas nécessaire d’identifier tous les fragments tryptiques pour valider l’identification d’une protéine.
En revanche, en peptidomique, le but ultime est d’identifier le maximum de peptides possible même s’ils dérivent du même précurseur protéique, car chaque peptide peut avoir une fonction distincte [9]. Les techniques protéomiques et peptidomiques partagent le problème de gamme dynamique limitée de la plupart des spectromètres de masse par rapport à la gamme de concentration beaucoup plus large des protéines ou des peptides présents dans les échantillons biologiques. En général, les techniques de spectrométrie de masse (MS) peuvent aisément détecter des protéines et des peptides présents en quantité importante ou modérée mais s’adaptent mal à la détection des molécules présentes en faible quantité.
Contrairement à la protéomique, la gamme dynamique des analyses peptidomiques est souvent limitée par la dégradation des protéines. Dans des conditions standard de dissection des tissus et d’extraction des peptides, il y a une petite fraction de protéines qui se dégradent par des processus enzymatiques ou non. La plupart des études protéomiques ne seront pas affectées si une faible proportion de protéines est dégradée pendant que la majorité reste intacte. Cependant, les peptides bioactifs sont généralement présents en faible quantité par rapport aux protéines majeures. Ainsi la dégradation (même infime) de ces protéines majeures peut masquer les signaux plus faibles des peptides endogènes d’intérêt. Il est donc nécessaire de s’affranchir de cette limitation en choisissant par exemple des tissus pauvres en enzymes [10] ou alors en enrichissant les peptides sur des colonnes d’affinité.