Les différents types de transduction
L’hybridation de la cible sur la sonde immobilisée à la surface de l’électrode est suivie en réalisant des mesures électriques avant et après l’hybridation permettant à la fois la détection et la quantification du fragment d’ADN. La méthode choisie pour construire le biocapteur va également jouer un rôle primordial dans les performances de robustesse et de sélectivité du capteur. De plus, pour des raisons de coût et d’applicabilité du capteur au diagnostic médical, la transduction doit être réalisée sans marquage de la cible. Dans un premier temps, il est intéressant d’exposer les différentes techniques de transduction électrochimiques sans marquage de la cible afin d’en étudier les caractéristiques et de choisir celle qui semble correspondre le mieux aux contraintes imposées par ce projet. Les capteurs peuvent être classés dans 7 grandes familles selon la méthode de transduction adoptée. transduction se fait en mesurant la variation de la charge dans la couche dense de Helmoltz due à l’hybridation (Figure I-20). En effet, lorsqu’une électrode solide est immergée dans une solution, il se produit une diffusion d’espèces chargées au voisinage de l’interface électrode/électrolyte dans la couche de Helmoltz entraînant la formation d’une capacité interfaciale. Par la suite, toute variation au niveau de cette couche, comme l’hybridation d’une cible ADN sur la sonde, entraîne une variation de la capacité interfaciale. Cette variation de charge durant l’hybridation est due à l’augmentation de la densité de charges négatives, portées par les groupements phosphates des bases nucléotidiques, et de contre-ions (sodium par exemple) à la surface de l’électrode. Il se produit alors un effet de charge miroir menant à la variation de la densité de porteurs de charge dans le métal ainsi qu’à la variation de la capacité de la double couche électrochimique. Il a été montré que dans le cas d’une électrode métallique, la variation de potentiel se produit majoritairement dans la double couche électrochimique (Figure I-21 A) alors que dans le cas d’une électrode semi-conductrice, la variation de potentiel se produit majoritairement dans l’électrode (Figure I-21 B)[70].
Généralités
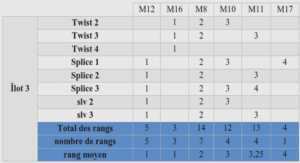
Cette méthode permet la détection de concentration beaucoup plus faible que l’oxydation de la guanine jusqu’à une LOD de 1× 10 [71] (Tableau I-2). Cette technique permet également de différencier une cible parfaitement complémentaire d’un fragment avec une unique non-complémentarité (mismatch). Cette méthode est donc très performante mais les mesures prennent jusqu’à plusieurs heures et elle n’est pas parfaitement adaptée au diagnostic puisque l’adsorption non spécifique d’espèces à la surface du capteur génère un bruit électrochimique important. De plus, les matériaux d’électrodes coûtent cher et il semble difficile de concevoir un capteur économique par cette méthode. plus précisément de la guanine contenue dans l’ADN. Les bases aromatiques des nucléotides sont assez sensibles à l’oxydation. La guanine est celle qui possède le potentiel d’oxydation le plus bas parmi les 4 bases. La réaction d’oxydation de la guanine a été étudiée en détail pour parvenir à proposer un mécanisme (Figure I-22)[77], [78]. Son potentiel est donné à 0,94 V .
Ces obstacles ont été surmontés en adoptant une stratégie intéressante mais plus coûteuse qui consiste à remplacer toutes les guanines de la sonde par des bases inosines (I)[80] (Figure I-24). L’inosine s’oxyde à un potentiel plus élevé que la guanine et n’est donc pas détectée lors de la mesure. Cela contribue à améliorer le ratio signal/bruit tout en rendant le capteur réutilisable. Cependant, l’inosine n’est pas complémentaire de la cytosine comme la guanine mais elle forme des complexes avec les trois autres bases par liaisons hydrogènes (Figure I-24). Cette modification permet donc une quantification plus précise, mais une sélectivité plus faible qui ne conduit pas à la détection d’un fragment à un seul mismatch si celui-ci est situé en regard d’une inosine. Il est possible de trouver dans la littérature des capteurs avec une limite de détection femtomolaire (10 ) mais les cibles sont alors des ADN génomiques fragmentés[81]. Dans ce cas, c’est le grand nombre de guanines présentes et oxydées qui permet d’atteindre cette limite. Cette méthode n’est donc pas applicable aux miRNAs. D’autre part, la meilleure limite de détection (LOD) obtenue pour des petits acides nucléiques par cette méthode est de 5 × 10 électro-active greffée sur la sonde peuvent également être fabriqués. En effet, une molécule ayant une activité rédox peut être greffée à l’extrémité de la sonde ADN comme le bleu de méthylène (MB) par exemple[86]. Il suffit ensuite de mesurer l’activité de cette molécule par électrochimie. Dans ce cas, deux configurations sont envisageables. Pour la première, la sonde est greffée en monocouche, mais repliée dans une configuration d’épingle à cheveux où la molécule électro-active est très proche de la surface de l’électrode. Pour la seconde, la configuration est plus classique puisque la sonde est greffée en monocouche, déployée à la verticale de l’électrode (Figure I-25).