L’évolution du rapport entre l’humain et ses génomes
La génomique définit l’organisation des génomes sur leur support, l’ADN (ou l’ARN chez les virus), ainsi que la manière dont ils régissent l’ensemble des mécanismes moléculaires comme l’expression d’ARN, ou la production de protéines dans les organismes. Cette discipline s’est progressivement immiscée dans la vie humaine dans les dernières décennies, aussi bien dans les domaines des professionnels de santé que de manière récréative dans le grand public. Par exemple, depuis 2019 et jusqu’à aujourd’hui en 2022, la pandémie de SARS-CoV-2 a vu croître le nombre d’informations génomiques délivrées au grand public, notamment sur l’existence de variants viraux et de leur impact sur l’infection . De plus, les années 2000 sont le début des tests génétiques « récréatifs », qui pour la plupart peuvent renseigner sur les ancestralités d’une personne en se basant sur les similarités avec plusieurs populations humaines. Bien qu’ils soient interdits en France , environ 100 000 à 200 000 y aurait recours annuellement . Ainsi, des entreprises comme 23andme ont même étendu leur rôle récréatif en obtenant l’autorisation de tester certaines pathologies.
Ces situations montrent l’augmentation de l’accès et de l’attrait de la population pour la génomique mais soulignent également la diversité des génomes étudiés. Le monde de la médecine et de la recherche connaît cette diversité. Ainsi les progrès continuels de la génomique humaine ont modifié le parcours de santé pour y incorporer des tests de pathologies génétiques ou évaluer la compatibilité en transplantation par le groupe sanguin ou les gènes du Human Leukocyte Antigen (HLA) . Malgré l’omniprésence de la génomique dans la recherche en santé, notamment dans l’étude des pathologies et la connaissance de l’hétérogénéité des génomes humains, l’étude de l’interaction entre les pathologies et les différences génétiques entre les populations reste minoritaire. Dans le cas de l’insuffisance rénale, la différence génétique des populations est intégrée dans certains scores de prédiction de la fonction rénale mais leur impact réel à long terme sur le traitement des patients est questionné .
Ainsi, la génomique semble prendre une importance de plus en plus grande pour comprendre les pathologies. Son inclusion de la diversité génétique humaine reste pourtant limitée par le manque de connaissance des séquences de populations non-européennes et par son utilisation dans les maladies complexes. Afin de mieux comprendre l’état actuel des connaissances en génomique, il est intéressant de se pencher brièvement sur les principes de bases de l’hérédité et de la génétique humaine pour évaluer comment les variations dans les populations humaines sont prises en compte.
Le Complexe Majeur d’Histocompatibilité (CMH), un génome de l’immunité dans un génome
Les génomes humains regorgent de diversité : des polymorphismes simples, mutations, insertions/délétions, ou changements structuraux ; à des fréquences différentes entre des populations géographiquement éloignées mais également au sein de populations proches. Cette diversité a un impact sur tous les traits pouvant affecter l’humain. L’exemple parfait de l’impact de la diversité génétique sur les humains est le Complexe Majeur d’Histocompatibilité (CMH). Le CMH est une région génomique située sur le chromosome 6 et son nom découle de sa découverte comme un facteur de compatibilité important lors de la transplantation.
La découverte du HLA en transplantation : Dès 1936, l’immunologiste Peter Gorer parvient à différencier des souris consanguines à partir des anticorps contenus dans du sérum de lapin, ces différences sont appelées types antigéniques car les anticorps reconnaissent des molécules nommées alors antigènes . Il faut cependant attendre 1958 pour que Jean Dausset , Rose Payne et Jon Van Rood identifient de manière concomitante des « iso-leuco-anticorps » permettant d’agglutiner les cellules du sang. Tout comme les différents groupes sanguins, ces facteurs semblent reconnus par des anticorps qui fixent les molécules considérées comme étrangères au corps. Plusieurs années de recherche et congrès immunogénétiques ont été nécessaires pour élucider l’organisation génétique de ces antigènes, nommés par la suite Human Leukocyte Antigen (HLA).
Il a été rapidement démontré que ces antigènes étaient essentiels en transplantation et, à l’instar des groupes sanguins, que le HLA était un facteur d’histocompatibilité (i.e. compatibilités des tissus entre eux). Lors de greffes de peau, Ceppellini et al., ainsi que Amos et al. ont démontré que le greffon était conservé plus longtemps entre des frères et sœurs qui partageaient leurs gènes HLA. En 1965, l’idée de l’importance de la correspondance HLA entre deux individus pour la greffe de rein a aussi émergé . Bien que la médecine actuelle, notamment l’amélioration des traitements immunosuppresseurs, a permis d’augmenter la survie du greffon entre donneurs et receveurs de HLA différents , le HLA reste un marqueur essentiel dans les greffes. La greffe de cellules souches hématopoïétiques, à l’origine de toutes les cellules sanguines, est nécessaire dans de nombreux cancers et maladies auto-immunes. Elle doit strictement suivre cette compatibilité HLA . La nécessité de trouver un donneur compatible HLA dans ce cas est toujours essentielle et des outils sont développés pour trouver ces donneurs . La dénomination d’antigène donnée au HLA vient de sa capacité à être reconnu par le système immunitaire, chargé de morceaux de protéines, ce qui mène à la génération d’anticorps contre lui. Plus tard, on trouvera sa fonction de présentation d’antigènes.
Les généralités de l’immunité humaine
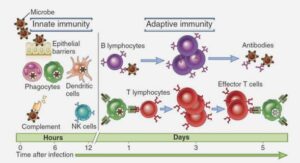
Le HLA a été identifié pour son rôle de carte d’identité de la cellule. La diversité d’allèles existants peut entraîner une reconnaissance de ces derniers par le système immunitaire, provoquant la production d’anticorps dirigés contre ces molécules lors de transplantations, chez les personnes enceintes, ou lors de transfusion fréquentes, on parle alors de rejet. Cependant, ces rejets sont une conséquence du rôle principal des molécules HLA dans la présentation d’antigènes, essentielle à l’immunité adaptative. L’immunité humaine correspond à l’ensemble des cellules et de leurs actions qui constituent les mécanismes de défense face à tout corps étranger qui entre dans l’organisme. Les bactéries, les champignons, les parasites, les virus, les cellules cancéreuses ou les toxines, sont généralement considérés comme les principales causes biologiques d’une réaction immunitaire . L’immunité apporte une réponse immédiate et non-spécifique, dite innée, à l’encontre de ces corps étrangers grâce aux barrières physico-chimiques naturelles ainsi qu’à certaines cellules comme les macrophages ou les cellules Natural Killer (NK) . Une immunité adaptative est également mise en place après environ 7 jours, le temps nécessaire pour que les lymphocytes B et T reconnaissent spécifiquement des pathogènes et puissent se multiplier. Une mémoire immunitaire se construit à la suite de cette première rencontre avec un pathogène, ce qui permettra une réponse plus rapide à l’avenir . Ces deux parties ne sont pas indépendantes et interagissent en s’activant l’une et l’autre .
Le HLA intervient de manière centrale dans l’immunité adaptative car ce sont ces molécules qui présentent les antigènes, les molécules potentiellement délétères, aux lymphocytes T. Chacun des lymphocytes T dispose d’un gène qui code pour un T-Cell Receptor (TCR), réarrangé différemment, qui va reconnaître spécifiquement un antigène, un couple HLA-peptide de l’individu. Cet ensemble forme un large répertoire capable de reconnaître virtuellement tous les pathogènes . Deux grandes catégories de molécules HLA vont ainsi stimuler le système immunitaire adaptatif par le biais de ces TCR : les HLA de classe I et ceux de classe II, nommés ainsi car ils sont situés dans deux loci différents du CMH.
Les HLA de classe II alimentent la réponse immunitaire innée
La structure et la fonction générale du HLA de classe II : Les HLA de classe II jouent un rôle similaire de présentation d’antigènes à celui des HLA de classe I.
Cependant, les peptides présentés sont exogènes, c’est-à-dire d’abord phagocytés par la cellule. Les molécules HLA de classe II sont ainsi exprimées par des cellules présentatrices d’antigène : des cellules de l’immunité innée, comme les cellules dendritiques, les monocytes, et les macrophages ; des cellules de l’immunité adaptative, les lymphocytes B ; et les cellules épithéliales, mais en condition d’inflammation seulement .
La structure des molécules HLA de classe II est semblable à celle des molécules de classe I au niveau du sillon peptidique , cependant elle diffère d’un point de vue plus global . En effet, les molécules de HLA de classe II sont des hétérodimères formés de deux molécules HLA codées par des gènes différents avec une partie α et une partie β (ex. HLA-DRA1 et HLA-DRB1). Ce sont les domaines α1 et β1 des deux molécules qui vont créer le sillon peptidique, ouvert de part et d’autres pour accommoder des peptides de taille variable. Seuls les lymphocytes T CD4+ interagissent avec les HLA de classe II par le biais de leur TCR et de leur corécepteur CD4 : l’activation entraîne une modification de leur profil d’expression pour devenir des lymphocytes T Helper (Th). Ces lymphocytes Th disposent d’un phénotype différent selon le cocktail de cytokines présent dans leur environnement lors de l’activation. Deux types majeurs, Th1 et Th2, Le HLA de classe II est constitué de deux molécules de HLA différentes α et β qui s’associent pour former le sillon peptidique. Le polymorphisme caractéristique des molécules HLA est le plus souvent restreint à la partie β de la molécule. Traduit d’un template de biorender.com. favorisent les réponses cellulaires et humorales respectivement . D’autres populations ont été découvertes comme les Th17 qui sont spécialisés dans l’inflammation des tissus, ou les Tregs qui ont la capacité de réduire la réponse immunitaire et même d’atteindre la tolérance par homéostasie immunitaire.
Le chargement des peptides : De la même façon que pour le HLA de classe I, les molécules HLA de classe II sont traduites dans le RE. Cependant elles en sortent rapidement en se liant à la chaîne invariante Ii (Figure II-5, traduit et adapté d’un template biorender.com). Cette protéine dispose d’un domaine CLIP (Class II-associated invariant chain peptide) qui remplit le rôle du peptide en se logeant dans le sillon peptidique . Une vésicule contenant les molécules de HLA de classe II fusionne alors avec un endolysosome, issu de la phagocytose de protéines externes par la cellule, pour devenir un endosome tardif, le compartiment HLA classe II, MIIC . Les protéases contenues dans l’endolysosome dégradent ainsi les protéines phagocytées mais également une partie de la chaîne invariante, laissant la partie CLIP dans le sillon peptidique. La molécule HLA-DM, à l’instar de TAP pour les HLA de classe I, va servir de chaperonne, facilitant l’échange de CLIP pour des peptides de plus forte affinité, à savoir les peptides étrangers, permettant ainsi l’adressage à la membrane d’un complexe CMH-II/peptide du non-soi .
Le processus diffère dans les lymphocytes B où un hétérodimère HLA-DM/HLA-DO modifie le répertoire peptidique en limitant l’accès de certains peptides (85). Ce répertoire peptidique est néanmoins très différent des HLA de classe I car le sillon peptidique de la molécule est plus large et ouvert de chaque côté. Les peptides accommodés sont généralement plus grands, de 12 à 25 acides aminés .
La situation génomique des gènes HLA
L’ensemble du système HLA est situé au niveau du locus 6p21, sur le bras court du chromosome 6 dans le CMH. Cette région est la plus dense du génome en terme de gènes, plus de 200, soit 1% de tous les gènes humains, alors qu’elle ne compte que pour 0,1% de la longueur du génome. La région du CMH s’étend classiquement sur 4 millions de bases nucléotidiques (Mb) du gène MOG au gène COL11A2 (29 657 002–33 192 499, GRCh38.p13). En tenant compte des motifs particuliers de déséquilibre de liaison de la région, il est possible de définir un CMH étendu (CMHx / xMHC en anglais) selon la définition proposée par Horton et al. (53). Cette région peut s’étendre sur 8Mb (25 726 063– 33 400 556, GRCh38.p13).
Comme présenté sur la Figure II-1, le CMH a été divisé en trois régions nommées classe I, II, et III. Aucun gène HLA n’est présent dans le CMH de classe III, ils sont ainsi divisés entre les classes I et II. La variété de structure et de fonctions HLA évoquée plus tôt correspond aussi à une localisation différente. L’ensemble des gènes du système HL est situé au début et à la fin du CMH, sur les deux brins d’ADN. Les molécules HLA de classe I sont un groupe hétérogène car elles sont traduites à partir de 6 gènes différents : HLA-A, HLA-B, HLA-C, HLA-E, HLA-F, et HLA-G. Les trois premiers gènes (-A, -B et -C) sont dits «classiques» car ils représentent les molécules HLA exprimées dans toutes les cellules et leurs produits interagissent avec le TCR et le corécepteur CD8 des lymphocytes T CD8+. Les trois derniers (-E, -F, et -G) sont dits « non-classiques » car ils ont des expressions et interactions spécifiques. Elles sont néanmoins toutes présentes sous forme d’hétérodimère en association avec une molécule de β-2- microglobuline, dont le gène est situé sur le bras long (15q21.1) du chromosome 15. Les HLA-E et -F sont ubiquitaires mais les cellules les expriment moins. De plus, ils n’interagissent pas avec des TCR (ex. le HLA-E interagit avec l’hétérodimère CD94 / NKG2A ou CD94 / NKG2C sur les cellules NK). Quant à la molécule HLA-G, elle est connue pour jouer un rôle de tolérance à l’interface entre le fœtus et l’utérus, en interagissant avec le CD8 des lymphocytes T et LILRB1, LILRB2 et KIR2DL4 chez les cellules NK.
Pour les molécules de classe II, les gènes HLA classiques sont au nombre de 12, avec : HLA-DRA, HLA-DRB1, HLA-DRB3, HLA-DRB4, HLA-DRB5, HLA-DQA1, HLA-DQA2, HLA-DQB1, HLA-DQB2, HLA-DQB3, HLA-DPA1, et HLA-DPB1. Ce sont également des hétérodimères, mais chaque chaîne (α et β) est encodée par un gène HLA (ex. les complexes HLA-DP sont obtenus à partir des produits de HLA-DPA1 et HLA-DPB1). Le cas de la chaîne β de la molécule HLA-DR est particulier puisque plusieurs gènes DRB peuvent s’associer avec DRA. DRB3/4/5 ont une variation dans leur nombre de copies (copy number variation, CNV), ce sont des variants structurels et peuvent être présents ou absents ; seul le gène DRB1 est présent dans tous les cas. L’organisation génomique de ces gènes peut être représentée en plusieurs haplotypes, qui peuvent être statistiquement liés à certains allèles de DRB1). Ce locus reste encore aujourd’hui un des défis de la compréhension des loci HLA.
L’historique de l’élucidation des allèles HLA
Le typage HLA est l’identification des allèles HLA portés par un individu. Il est aussi appelé génotypage HLA, mais contrairement aux puces de génotypage qui informent sur un polymorphisme unique, celui-ci en indique plusieurs. Aujourd’hui, le séquençage HLA est le standard de typage, cependant, il a fallu plusieurs dizaines d’années avant de pouvoir connaître exactement l’organisation ainsi que la séquence exacte des gènes HLA.
Dans les dix dernières années, le nombre d’allèles a quadruplé, grâce aux changements des technologies de séquençage et leur application au HLA.
La sérologie, l’interaction anticorps-HLA : À l’origine, l’ensemble du HLA était connu comme un unique antigène découvert en 1958 par Jean Dausset . L’ajout du sérum d’un patient à un extrait de moelle osseuse d’un autre a mené à l’agglutination des lymphocytes présents , une réaction analogue à celle entre des individus de groupes sanguins différents. Cependant, les individus testés étaient de même groupe sanguin, indiquant la présence d’un antigène différent entraînant cette réaction. Celui-ci a été nommé MAC, d’après les initiales des patients possédant l’anticorps contre cet antigène . L’agglutination leucocytaire a été observée sur près de 60% des patients, et l’existence de cet antigène a rapidement impliqué celle d’autres antigènes similaires . Dans les années suivantes, plusieurs techniques basées sur cette réaction ont servi pour identifier l’ensemble des antigènes HLA, dont les tests de microlymphocytotoxicité par Terasaki et al. Cette technique repose sur des tests systématiques d’anti-sera ou d’anticorps monoclonaux connus sur des lymphocytes T et B de patients (pour le HLA de classe I) ou de lymphocytes B (pour le HLA de classe II). Les anticorps interagissent avec les molécules HLA correspondantes, puis un lavage élimine les anticorps qui n’interagissent pas. En ajoutant un sérum de lapin en guise de complément, celui-ci est activé par le fragment Fc des anticorps, les cellules sont ainsi lysées. En ajoutant un fluorochrome qui s’illumine seulement en présence de l’ADN libéré par la lyse puis en comparant les résultats entre les différents anticorps, il est possible d’inférer les allèles HLA de l’individu . Cependant, la complexité de cette méthode, additionnée aux résultats mitigés obtenus lors de son application en transplantation l’ont vue remplacée par les techniques moléculaires.
Table des matières
I – L’EVOLUTION DU RAPPORT ENTRE L’HUMAIN ET SES GENOMES
I.1 – D’une vision péremptoire de la génétique mendélienne aux multiples intrications de la génétique polygénique
I.2 – L’impact de la diversité génétique et les limites actuelles de son étude
II – LA DIVERSITE DU HLA ET SON INTRICATION DANS LA BIOLOGIE HUMAINE
II.1 – Le Complexe Majeur d’Histocompatibilité (CMH), un génome de l’immunité dans un génome
II.1.1 – La découverte du HLA en transplantation
II.1.2 – Le HLA est l’antigène qui cache la forêt
II.2 – Le système HLA est la clef de voûte du système immunitaire adaptatif
II.2.1 – Les généralités de l’immunité humaine
II.2.2 – Les HLA de classe I veillent à l’intégrité de toutes les cellules
II.2.2.1 – La localisation et fonction générale des HLA de classe I
II.2.2.2 – Le chargement des peptides
II.2.3 – Les HLA de classe II alimentent la réponse immunitaire innée
II.2.3.1 – La localisation et fonction générale du HLA de classe II
II.2.3.2 – Le chargement des peptides
II.3 – La description des gènes et de la diversité du HLA : un marathon génomique et technologique
II.3.1 – La situation génomique des gènes HLA
II.3.2 – Les allèles HLA, ou la complexité par le nombre
II.3.3 – L’historique de l’élucidation des allèles HLA
II.3.3.1 – La sérologie, l’interaction anticorps-HLA
II.3.3.2 – La PCR-SSO / PCR-SSP, obtenir un allèle polymorphisme par polymorphisme
II.3.3.3 – La PCR-SBT, les premières séquences complètes
II.3.3.4 – Le Next-Generation Sequencing (NGS)
II.3.4 – La nomenclature HLA
II.3.4.1 – Les différentes résolutions HLA
II.3.4.2 – La terminologie du HLA en dehors de la nomenclature actuelle
II.4 – La diversité populationnelle des gènes HLA et leurs origines évolutives
II.4.1 – Les bases de données de la recherche HLA
II.4.2 – La diversité au-delà de la multitude des allèles
II.4.2.1 – Le sillon peptidique est le foyer des polymorphismes HLA
II.4.2.2 – Les allèles HLA ont une répartition inégale en population 4
II.4.2.3 – Des fréquences qui varient différemment selon les ancestralités
II.4.3 – L’origine évolutive du CMH et de la diversité des gènes HLA
II.4.3.1 – Une région ancestrale partagée par le règne animal
II.4.3.2 – Une diversité d’hypothèses pour comprendre le polymorphisme HLA
III – LES ASSOCIATIONS GENETIQUES AVEC LES PATHOLOGIES : LE REVERS DE LA MEDAILLE DU HLA
III.1 – L’association génétique dans la région du CMH
III.2 – Une protection efficace des populations contre les infections, mais faillible à l’échelle individuelle
III.2.1 – Les associations majeures du HLA avec des pathologies infectieuses
III.2.2 – Les associations élusives de la pandémie de SARS-CoV-2
III.3 – Les maladies auto-immunes : un excès de zèle immunitaire ? L’exemple de la sclérose en plaques (SEP)
III.4 – Des exemples de la diversité des associations dans le CMH
IV – UNE GRANDE DIVERSITE IMPLIQUE DE GRANDES DIMENSIONS : QUAND LA STATISTIQUE ET L’INFORMATIQUE SE METTENT AU SERVICE DE LA GENETIQUE
IV.1 – Les différentes facettes de l’analyse bioinformatique du HLA
IV.1.1 – Les réponses in silico sont en première ligne pour étudier le HLA
IV.1.1.1 – Les corrélations HLA-trait et leurs multiples failles
IV.1.1.2 – Sur la piste des allèles HLA à risque et protecteurs grâce à l’exploration du peptidome
IV.1.2 – L’association statistique dans la région du CMH
IV.1.2.1 – Le CMH est le berceau de milliers d’associations génétiques emmêlées dans des motifs de déséquilibre de liaison
IV.1.2.2 – Les associations HLA lient directement les traits et les allèles
IV.1.3 – L’extension des études immunogénétiques à partir du HLA
IV.2 – Les données manquantes en HLA : contourner l’absence d’information par le contexte génétique
IV.2.1 – Les concepts généraux de l’imputation de données
IV.2.2 – L’imputation SNP, une application statistique de grande envergure en génomique
IV.2.3 – L’inférence statistique HLA, vers l’immunogénétique pour tous
IV.2.3.1 – La déséquilibre de liaison de la région du CMH, un faisceau d’indice pour retrouver l’identité d’un allèle HLA
IV.2.3.2 – Le séquençage HLA : une solution infaillible ?
IV.2.3.3 – Les limites de l’inférence statistique
IV.3 – L’exploration de la structure génétique des populations humaines et la répartition de la diversité
IV.3.1 – Les distances génétiques, de la parenté à l’ancestralité
IV.3.1.1 – La quantification de la proximité génétique entre des individus ou des populations
IV.3.1.2 – La diversité génétique individuelle à travers le prisme des générations
IV.3.2 – Les méthodes de réduction de dimensions et leurs utilisations en génomique
IV.3.2.1 – La construction d’un nouvel espace avec l’ACP
IV.3.2.2 – L’UMAP et la topologie des données comme point de repère
V – PROBLEMATIQUE ET OBJECTIFS DE LA THESE
VI – NAVIGUER LES EAUX TROUBLES DE L’IMPUTATION HLA AVEC LE SNP-HLA REFERENCE CONSORTIUM (SHLARC)
VI.1 – Un projet international pour démocratiser les études d’association HLA
VI.1.1 – La création d’un environnement propice à l’imputation HLA
VI.1.1.1 – CAAPA, un consortium pour la diversité génétique africaine
VI.1.1.2 – La création de modèles d’imputation HLA avec HIBAG
VI.1.1.3 – Le façonnement d’un nouvel environnement pour faire prospérer l’imputation HLA
VI.1.2 – Article – SNP‐HLA Reference Consortium (SHLARC) : le partage de données HLA et SNP dans le but de promouvoir les analyses génomiques centrées sur la région du CMH
VI.2 – L’amélioration de l’imputation HLA dans des populations sous-représentées
VI.2.1 – Le contournement du manque de diversité par une nouvelle méthodologie
VI.2.1.1 – Les variations de fréquences HLA dans les populations et leur impact sur l’imputation
VI.2.1.2 – Le changement de métrique d’imputation HLA
VI.2.2 – Article – Explorer l’imputation HLA des populations d’ancestralité composite avec la réduction de dimension
VI.3 – La mise à profit des données génétiques et des outils bioinformatique pour explorer extensivement le rôle du HLA
VI.3.1 – L’association HLA à la maladie de Parkinson change selon le statut tabagique
VI.3.2 – Article – L’interaction revisitée entre HLA-DRB1 et le tabagisme dans la maladie de Parkinson
VII – DISCUSSION
VII.1 – La trajectoire de l’imputation HLA
VII.1.1 – La recherche de pistes d’amélioration par la génération de nouvelles données
VII.1.2 – La création de panels de références personnalisés et ses limites
VII.1.3 – Le nouveau monde de l’imputation HLA
VII.2 – L’évolution de l’analyse HLA et son impact sur le monde de la génomique et de la clinique
VII.2.1 – De l’association génétique des SNP du CMH à une analyse complète du HLA
VII.2.2 – Quel futur pour l’imputation HLA ?
VII.2.3 – Les conséquences globales des avancées technologiques en immunogénomique
VIII – CONCLUSION
IX – REFERENCES BIBLILOGRAPHIQUES
X – LISTE DES COMMUNICATIONS SCIENTIFIQUES
X.1 – Publications
X.2 – Communication orales
X.3 – Posters
X.4 – Autres communications