Evaluation de la segmentation semi-automatique du logiciel Brain
Nous avons constitué une base de données de 20 images de patients différents (Tableau 13). Ces images sont acquises avec des séquences de type TSE pondérées en T2 (TR et TE allongés) à 1,5 T, dans le plan axial, passant par les noyaux gris centraux et les ventricules latéraux. L’épaisseur de coupe est de 2 à 4 mm, la taille des pixels est variable (de 0,39 x 0,39 à 0,70 x 0,70). Afin de valider la robustesse de l’algorithme, les images proviennent de différentes IRM réalisées à terme chez d’anciens nouveau-nés prématurés. Pour chacune de ces coupes, un observateur senior a manuellement segmenté les différentes structures cérébrales : liquide cérébro-spinal péricérébral, noyaux gris centraux, ventricules latéraux, substance grise et substance blanche, en y recherchant d’éventuelles hyperintensités. Un second observateur senior a également détecté et segmenté les hyperintensités de la substance blanche (Illustration 41). segmentations A et B, le coefficient de similarité de Dice est défini par 2 |A∩B| / (|A|+|B|). Cet indice est normalisé : 0 indique une dissemblance complète ; 1 indique un accord complet. Le logiciel Brain a été utilisé avec les paramètres par défaut. Seulement dans quelques cas, le seuil d’incorporation du contenu intracérébral a été abaissé de 0,3 à 0,2 afin d’inclure toutes les régions d’intérêt. Quelques ajustements manuels ont été effectués pour inclure ou exclure les zones détectées de substance grise ou blanche par défaut ou excès respectivement. Les coefficients de similarité de Dice ont été calculés entre les segmentations réalisées manuellement par l’observateur senior et celles obtenues avec le logiciel Brain, puis entre deux segmentations du logiciel Brain.
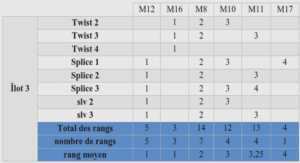
Afin de valider la segmentation semi-automatique (Illustration 42), les résultats de segmentation du logiciel Brain sont comparés à la segmentation manuelle, qui sert de référence. Le calcul du coefficient de Dice permet de quantifier cette similarité, selon les structures cérébrales. La comparaison de deux segmentations successives du logiciel Brain permet d’étudier la reproductibilité et la robustesse de l’algorithme, même lorsque l’opérateur a effectué des corrections. Le tableau 14 permet de quantifier les disparités de segmentation entre les différentes structures cérébrales. Les structures simples avec un contraste marqué comme les noyaux gris centraux ou les ventricules latéraux ont des coefficients de similarité bons, ce qui témoigne d’une bonne efficacité de l’algorithme. Les structures dont les contours sont plus sinueux, avec un volume partiel beaucoup plus important, telles que la substance grise corticale et la substance blanche, ont des coefficients de similarité moins bons entre l’algorithme du logiciel Brain et la segmentation manuelle. En revanche, les deux segmentations semi-automatiques successives réalisées avec le logiciel Brain ont des coefficients de similarité excellents, bien meilleurs que ceux obtenus par comparaison avec la segmentation manuelle, quelles que soient les structures étudiées. La reproductibilité de la segmentation des structures cérébrales néonatales est donc nettement améliorée grâce au logiciel Brain.
Résultats sur un échantillon d’images obtenues à 3 T
Afin d’évaluer la capacité du logiciel Brain à segmenter les différentes coupes passant par l’étage sus- tentoriel, nous avons comparé les résultats obtenus de façon semi-automatique aux résultats d’une segmentation manuelle d’un observateur senior. Les trois patients sont d’anciens grands prématurés, dont l’IRM a été réalisée à l’âge du terme théorique. Pour chaque structure visualisable ont été calculés, coupe par coupe, les coefficients de similarité de Dice. Le tableau contient les moyennes de ces coefficients (Tableau 15). Les coefficients de similarité de Dice sont bons pour la détection des noyaux gris centraux ainsi que des ventricules latéraux et de la substance blanche. Le coefficient de similarité de Dice est moyen pour la substance grise corticale, mais cela peut être expliqué par la difficulté de réalisation de la segmentation manuelle sur les images à 1,5 T, dont le volume partiel peut compliquer la délimitation. Afin de valider la qualité de l’algorithme du logiciel Brain sur des images acquises avec un champ magnétique de 3 T, nous avons utilisé une séquence pondérée en T2 composée de 50 coupes acquise chez un nouveau-né prématuré de 33 SA, nommée « Training » provenant du consortium européen Neonatal Estimation Of Brain Damage Risk And Identification of Neuroprotectants (NEOBRAIN) (Išgum et al., 2015). Les coupes sont de 2 mm d’épaisseur, la résolution de 0,35 x 0,35, la matrice de 512 x 512. Le contraste est spontanément plus marqué à 3 T que sur les image à 1,5 T (Illustration 43).