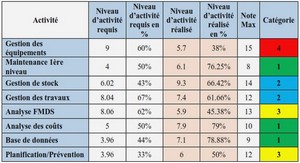
Études des populations relatives des AOB et NOB en eaux usées
Cette section synthétise les résultats des différents travaux de recherche traitant de l’étude des populations relatives d’AOB et NOB dans un contexte d’épuration des eaux usées et en particulier lors du traitement par biofiltration.
Enlèvement de l’azote par les filtres biologiques (BAF)
Les travaux réalisés par Pramanik et al. (2012) ont montré que les filtres biologiques aérés avaient la capacité d’être plus efficaces que les boues activées pour l’enlèvement de l’azote. D’ailleurs, Gieseke et al. (2003) rapportent que les NOB sont beaucoup plus sensibles que les AOB lors de baisses de l’oxygène dissous, de chocs toxiques, de variations de pH et de température lorsque le milieu de culture est en suspension, d’où l’importance d’assurer un contrôle étroit dans des procédés de type boue activée. La littérature scientifique rapporte peu d’études sur l’efficacité des filtres biologiques aérés, surtout en ce qui a trait à des applications en conditions réelles de traitement des eaux usées, et plus spécifiquement au niveau de l’enlèvement de la charge azotée par rapport à la présence relative des bactéries. De ce fait, Li et al. (2016) rapportent que les seules corrélations établies entre la population bactérienne et l’efficacité du traitement ont été observées en laboratoire (échelle pilote). La relation entre le potentiel de nitrification d’un bioprocédé et l’estimation de la population de bactéries nitrifiantes demeure à être étudiée.
Marqueurs moléculaires pour l’étude des AOB et NOB
Afin d’étudier la population des principaux groupes bactériens responsables de la nitrification au sein d’un biofilm, des techniques d’analyse basées sur les séquences génétiques, tel que la PCR ou la qPCR ou PCR en temps réel, ont été mises au point. Les séquences d’ARN ribosomal 16S ont été largement utilisées en tant que marqueur moléculaire en écologie microbienne en raison de leur caractère essentiel au fonctionnement bactérien, et dû au fait que cette séquence génétique se retrouve dans toutes les bactéries ainsi que de leurs propriétés évolutives (Case et al., 2007). Cependant, l’utilisation de ce marqueur peut parfois conduire à une mauvaise interprétation, car plusieurs copies de ce gène présentant des différences de séquence peuvent être présentes au sein d’un même organisme. Afin de corriger ce problème lors de l’étude des bactéries AOB, les travaux de McTavish et al (1993) ont proposé l’utilisation du gène codant la sous-unité A de l’ammonium monooxygénase (amoA). Ce gène est ubiquitaire à toutes les bactéries nitritantes et est spécifique à chaque espèce d’AOB. Ce gène est également présent chez les AOA. En ciblant un gène qui est associé à une caractéristique métabolique spécifique comme marqueur moléculaire pour des groupes de bactéries distincts, la précision de la méthode s’en trouve ainsi grandement améliorée. Dionisi et al. (2002) ont utilisé le gène amoA lors d’essais de PCR compétitive afin d’évaluer la présence relative des AOB au sein d’une biomasse en suspension. Ces travaux ont conduit à une amorce génétique spécifique à une espèce précise de Nitrosomonas et toutes les estimations de présence relative des AOB ont été fondées sur cette espèce. Les chercheurs ont par ailleurs estimé que les Nitrosomonas présentent deux copies du gène amoA par génome.
Dans le même ordre d’idée, afin d’étudier la présence relative des bactéries NOB, les travaux de Pester et al (2014) et Gruber-Dorninger (2015) ont suggéré l’utilisation du gène codant la sous unité B de l’enzyme « nitrite oxido-reductase » (nxrB). Ce gène est présent chez toutes les NOB et est nécessaire pour la métabolisation des nitrates à partir des nitrites. Plusieurs copies de ce gène sont présentes au sein des différentes espèces bactériennes du groupe des NOB. Ces mêmes chercheurs ont déterminé que le nombre de copies de ce gène variait de deux à six pour un large spectre d’espèces de NOB. Ainsi, en regroupant un maximum de NOB, l’estimation à quatre copies du gène nxrB par génome bactérien permet d’englober en moyenne toutes les espèces de NOB présentes dans un biofilm hétérogène complexe.
Relation entre AOB, NOB et la nitrification
Tel que démontré par Graham et al. (2007), la relation entre AOB et NOB pour la nitrification est imprédictible, mais une corrélation entre les deux a été clairement définie. Ces chercheurs ont émis l’hypothèse que les deux groupes bactériens devaient croître en collocation dans le biofilm afin de faciliter les échanges métaboliques. Lors d’essais menés par Dionisi et al. (2002) et Harms et al. (2003) sur les populations relatives des bactéries nitrifiantes, leurs observations ont montré que la population d’AOB semble être inférieure aux NOB. Toutefois, aucun abattement de nitrification a été rapporté en lien avec les populations présentes. Ainsi, la présence des bactéries nitrifiantes uniquement n’est pas garante de l’efficacité du traitement projeté. Par contre, plusieurs études ont rapporté que lors d’une nitrification fonctionnelle, la population relative de NOB devrait être logiquement supérieure à la présence relative des AOB (Daims, Nielsen, Nielsen, Schleifer & Wagner, 2001; Dionisi et al., 2002; Gieseke et al., 2003; Harms et al., 2003). Ce sujet reste toutefois à être étudiée plus en profondeur afin de confirmer cette relation .
Différentes études ayant utilisé des outils moléculaires afin d’assurer le suivi du développement des bactéries nitrifiantes ont rapporté une hausse des NOB par rapport aux AOB. Ainsi, les travaux de Dionisi et al. (2002) portant sur une station d’épuration par boues activées ont rapporté un nombre 62 fois plus important de Nitrospira sp. (NOB) par rapport aux cellules N-oligotropha (AOB). Quant aux travaux de Shramm et al. (1999) portant sur un réacteur nitrifiant à lit fluidisé, la population d’AOB observée tendait à décroître en fonction de la profondeur tandis que la population des NOB s’accroissait. Les travaux de Gieseke et al. (2001) portaient sur l’évaluation de la population des nitrifiantes d’une biomasse en suspension dans un réacteur biologique séquentiel (RBS). Leurs observations rapportent une hausse des NOB dans le premier 100 µm du biofilm d’environ 30 fois supérieures à la population d’AOB. Ces différents travaux semblent identifier comme cause probable le temps de rétention hydraulique (HRT) ainsi que la charge C/N afin d’expliquer la domination des NOB au sein d’un biofilm nitrifiant fonctionnel.
La polyvalence du métabolisme des NOB
La polyvalence du métabolisme des NOB particulièrement associé aux Nitrospira a été considérée afin d’expliquer leur supériorité dans le biofilm nitrifiant. Ainsi, dès 1986, Watson et al. (1986), avec la découverte d’une nouvelle espèce de bactérie chemolithoautotrophe capable d’oxyder les nitrites, la Nitrospira marina, émettaient l’hypothèse que ce groupe de bactéries pourrait être l’une des populations de NOB dominante dans l’environnement. Depuis, avec le raffinement des études métaboliques et génétiques, il a été démontré, entre autres par Lucker et al. (2010), que les Nitrospira constituent un groupe de bactéries nitrifiantes diversifié et parmi les plus répandus dans les écosystèmes naturels ainsi que dans les procédés biologiques des eaux usées. Les travaux de Koch et al. (2015) ont quant à eux mis en évidence une propriété métabolique inattendue chez les NOB du genre Nitrospira moscoviensis, une autre espèce répandue dans les eaux usées. Ces NOB ont un gène codant une uréase leur permettant ainsi de cliver l’urée présent en ammoniac. De ce fait, ces espèces ont la capacité d’alimenter en ammoniac les AOB responsables de la nitritation. Ainsi, Koch et al. (2015) proposent que cette alimentation réciproque avec les AOB et cette polyvalence métabolique puissent être des facteurs clés pouvant expliquer les observations sur l’ubiquité et la domination des NOB dans les systèmes naturels et les procédés de traitement biologique. Récemment, les travaux de recherche de Daims et al. (2015) ont mis en évidence la présence d’une espèce de Nitrospira, dont l’existence était autrefois hypothétique, capable d’accomplir le cycle complet de nitrification, soit les « comammox » (complete ammonia oxidizer). Le génome de cet organisme chemolithoautrotrophe encode une voie métabolique à la fois pour l’oxydation de l’azote ammoniacal et des nitrites pouvant être activée en même temps pendant la croissance. Une telle capacité remet en question le cycle de l’azote tel qu’on le connait et pourrait faire des comammox une espèce clé des procédés biologiques de traitement des eaux.
INTRODUCTION |