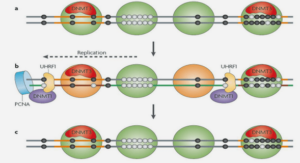
Cycle
La connaissance du cycle du paludisme permet de mieux comprendre les modalités de transmission.
Ainsi le paludisme est transmis, pendant la nuit, par la piqure d’un moustique appelé anophèle femelle. Le cycle se déroule successivement chez l’homme et chez l’anophèle. D’abord, chez l’homme où s’effectue la totalité du cycle asexué ou schizogonique (voir figure 1), nous avons deux phases : la phase hépatique ou pré-érythrocytaire qui correspond à la phase d’incubation, cliniquement asymptomatique et la phase sanguine ou érythrocytaire qui correspond à la phase clinique de la maladie. Ensuite, chez le moustique où s’effectue le cycle sexué ou sporogonique (voir figure 1). Lors du repas sanguin sur un sujet infecté, l’anophèle absorbe des gamétocytes qui se transforment en gamètes mâles et femelles. Ces gamètes fusionnent en un oeuf libre, mobile appelé ookinète. Cet oeuf pénètre par la suite la paroi stomacale du moustique pour devenir un oocyste sphérique produisant des centaines de sporozoïtes due à la division multiple du noyau. Les sporozoïtes sont ensuite relachés qui migrent jusqu’aux glades salivaires d’où peuvent, lors d’un repas sanguine, infecter un nouvel hote humain (Talma et al ; OMS, 2003).
Diagnostics moleculaire du paludisme
Le Dr Eniyou Cheryll Orierio de « MRCG unit the Gambia » a identifié avec succès une nouvelle cible, avec une très forte sensibilité dans les diagnostics moléculaires du paludisme qu’elle l’optimisa grace à une méthode d’amplication isotherme connu sous le nom de l’ancronyme LAMP (Loop Mediated Isothermal Amplification) (Umberto, 2016). Le diagnostic moléculaire permet une meilleure identification de l’espèce parasitaire lors et il est recommandé dans les cas de parasitémies très faibles. Il s’agit d’une ampliation génique à partir d’un échantillon de sang prélevé. Les avantages de cette nouvelle techniques sont : son extrême sensibilité, sa très bonne spécification et sa durée de réalisation qui très adaptée au diagnostic d’urgence (Bio67info, 2017).
Définition
Le polymorphisme nucléotidique (PN) ou polymorphisme d’un seul nucléotide (PSN) (singlenucléotide polymorphism en anglais (SNP)) est, en génétique, la variation d’une seule paire de base dans le génome entre les individus de la même espèce. On parlera généralement de forme allélique.
On a les SNPs bi-alléliques (bSNPs) et les SNPs multi-alléliques (mSNPs).
Un SNP bi-allélique est défini comme étant une position où on a un allèle de référence et un seul allèle alternatif. Par-contre, on parle de SNP multi-allélique si sur une même position du génome, on a plusieurs allèles alternatifs.
Les indels sont soit des insertions, soit des délétions (d’où le nom d’indels), c’est-à-dire l’ajout/diminution de plusieurs nucléotides.
Structure des données
Les données de génotypage, issues du « variant calling », c’est-à-dire processus d’identification de toutes les variations par rapport au génome de référence (Pf3D7_v3), sont stockées dans des fichiers VCF (Variante Calling Format). Ces types de fichiers donnent des informations sur les différences notées entre le génome de référence et celui des individus étudiés. En d’autres termes, ils récapitulent pour chaque chromosome, les positions où le génome de l’hôte diffère du génome de référence, l’allèle de référence et l’allèle mutant, la confiance qu’on porte sur le fait que c’est une vraie mutation (le nombre de reads soutenant l’allèle considéré), les différents génotypes identifiés sur les individus étudiés, et plein d’autres informations. (http://vcftools.sourceforge.net/man_latest.html , 12/02/2020).
Lieu de collection des échantillons
Les échantillons proviennent de 13 localités dont 03 au Sénégal et 10 en Gambie. Ces échantillons ont été collectionné par une équipe basé au MRC unit the GAMBIA dont le dirigeant est le professeur Alfred NGWA. La répartition géographique des 13 sites de collection est illustrée par la figure 3 cidessous.
Le tableau suivant résume le nombre d’échantillons collectés par année dans chaque localité.
Vérification de la qualité de séquençage
Cette étape consiste à filtrer les variantes sur la base des critères (MAF <5%, MQ ≥ 30, VQSLOD ≥2) établis pour s’assurer qu’ils ne proviennent pas d’erreurs de génotypage et / ou d’autres artefacts.
Le mapping quality (MQ) et le quality Score (QS) sont parmi les paramètres utilisés pour vérifier la qualité du séquençage. Le mapping quality est la certitude que la séquence lue est correctement alignée sur le génome de référence. Le quality Score (VQSLOD), en revanche, est la qualité de la correspondance entre les deux séquences, c’est-à-dire entre la séquence lue et la séquence de référence. (Robert P. Adelson et al, 2019). Cependant, MQ et QS ont été extraits sur les SNPs (bSNPs et mSNPs) / INDELs pour chaque chromosome. Donc pour une meilleure compréhension,
Nous avons visualisé ces différentes valeurs par des nuages de points (voir figure s1 au niveau de l’annexe).
Distribution des mSNPs et Indels sur le génome
L’un des facteurs faisant la robustesse d’une analyse de données génomique est le nombre de variantes utilisées. Ainsi, plus on a des SNPs (respectivement des Indels), plus les conclusions issues de l’interprétation des résultats de l’analyse son fiables. Cependant, il faut noter que lorsque les variantes ne sont pas équitablement dispersées sur tout le génome, cela peut engendrer des biais et donc une interprétation erronée des résultats. Pour s’assurer si les variantes utilisées couvrent l’ensemble du génome du parasite, nous avons dans un premier temps conçu le caryogramme de mSNPs et indels (Figure 5A). Puis, dans un second temps nous avons divisé chaque chromosome en différents intervalles de 5000 paires de base avec un chevauchement de 2500 paires de base afin de savoir si les variantes sont uniformément distribuées à travers les différents chromosomes. Par exemple : [0, 5000], [2500, 7500], [5000, 10000] … Ainsi pour chaque intervalle nous avons estimé le nombre d’INDELs et mSNPs. Ceci a permis d’avoir une idée globale de la densité de chaque variante le long des 14 chromosomes (figure 5B).
Proportion des variantes sur les différents gènes
Après la vérification de la qualité des variantes, nous avons identifiés pour chaque position (SNPs ou INDELs) le gène correspondant. Ainsi, nous avons déterminé le nombre de gènes couvrant les bSNPs,mSNPs et Indels (figure 6A et 6B). De plus, nous nous sommes intéressés au pourcentage de SNPs et indels couverts par des gènes connus comme étant impliqués à la résistance du parasite aux médicaments (figure 6C).
NB : les outils utilisés dans cette partie sont bcftools version 1.9 et des scripts écrits en python version 3.9.0 pour le traitement de ces données
Les nouvelles variantes
Définition
Une nouvelle variante est définie comme étant une mutation qui a été détectée à l’an N et qui n’existait pas à l’an N-1 ou bien à l’année de référence (Redmond et al, 2018).
Ainsi, nous avons d’abord considéré 1984, l’année d’introduction de la Chloroquine en tant que traitement de première ligne, comme année de référence et nous avons identifié par la suite les variantes qui ont existé au cours des autres années et non en 1984. Ceci permet d’identifier les mutations qui ont émergé sur le génome du parasite suit à l’introduction de la Chloroquine.
Enfin, afin de mieux comprendre à partir de quelle année certaines variantes sont apparues sur le génome du parasite, pour chaque paire d’années consécutives, nous avons identifié les variantes qui ont été décelées au cours d’une année et non pendant l’année précédente.
Les nouvelles variantes : comparées à 1984
Pour déterminer les nouvelles variantes comparées à 1984, les données ont été divisées en plusieurs groupes. Chaque groupe contient les échantillons collectés au cours d’une même année. Nous avons ensuite séparé les bSNPs, mSNPs et indels à partir de chaque groupe. Ainsi, nous avons comparé les positions des SNPs / INDELs de chaque chromosome à celles de 1984. Si une position est décelée en 1990, par exemple, et pas en 1984, alors elle est considérée comme étant une nouvelle variante. Voir figure 7A.
Après avoir obtenues les nouvelles variantes, nous avons calculé la fréquence de l’allèle alternatif sur chaque locus (SNPs / INDELs) de chaque année ce qui nous a permis d’avoir une vision globale. Voir figure 7B.
De ces fréquences, nous avons extrait les SNPs / INDELs dont leurs fréquences sont supérieures à 0,95 (voir figure s3A), celles qui augmentent (voir figure s3B) et celles qui fluctuent (voir figure s3C) entre 2000 et 2015. Enfin, pour chacune de ces trois catégories, nous nous sommes intéressés uniquement aux gènes impliqués dans la résistance en calculant le pourcentage de positions (SNPs ou INDELs) englobées par chaque gène par rapport au nombre total englobé par ces gènes (voir figure 7).
NB : Aucun SNPs ou indels dont leurs fréquences sont constantes ou diminues au cours du temps n’a été noté.
Les SNPs/INDELs qui sont en même temps rares et nouvelles au cours de temps comparées à 1984
Parmi les rares variantes déjà déterminées, nous avons cherché à identifier celles qui sont nouvelles par rapport à 1984 (figure 8). C’est ainsi grâce à des scripts écrits en python, nous avons déterminé le nombre de rares et nouvelles variantes, compare à 1984, pour chaque localité et année de collecte (voir figure s4 au niveau de l’annexe).
Les nouvelles variantes : pour chaque paire d’années consécutives
Précédemment, nous avons montré l’existence de nouvelles variations qui sont survenues sur le génome du Plasmodium falciparum à la suite de l’introduction de la chloroquine. Pour déterminer avec précision l’année à laquelle ces nouvelles variations ont été vues sur le génome du parasite pour la première fois, nous avons identifié les nouvelles variantes entre chaque paire d’année consécutive.
La figure 9A illustre les résultats obtenus.
Après l’extraction de ces nouvelles variantes, nous avons calculé pour chaque SNPs/INDELs la fréquence de l’allèle alternatif, relativement au nombre total de bSNPs (ou d’INDELs ou mSNPs respectivement), afin de savoir à quel point elles sont représentées dans la population étudiée. Voir figure 9B.
NB : Les outils utilisés dans cette partie sont bcftools et des scripts écrits en python pour le traitement de ces données.
Imputation des données manquantes et phasing
Lors de la création d’un fichier VCF, si la confiance portée sur l’existence d’une variante sur une position quelconque du génome d’un individu n’est pas assez grande, cette variante ne sera marquée comme étant génotypé. Elle est alors considérée comme une donnée manquante et représentée par « ./. ». Lorsqu’on a un fort taux de données manquantes, on peut penser que la plateforme de génotypage est défectueuse (Coulonges, 2011). Nous avons imputé (processus qui consiste à reconstruire les génotypes manquant sur la base d’informations utiles qui se trouvent dans la colonne FORMAT du fichier VCF) les données manquantes sur la base de l’AD (Allelic Depth). Si le nombre d’allèles supportant l’allèle de référence est plus grand, alors on le recode à 0 sinon on le recode à 1.
Le phasing consiste ici à recoder les positions hétéro-alléliques « 0/1 » par 0 ou 1. Chez les individus haploïdes, ce genre de génotype est causé lorsqu’il a une ambigüité entre l’allèle de référence et l’allèle alternatif ou lorsqu’on est en présence d’une infection multiple (différentes souches desparasites transmis à l’hôte suite à la piqûre de plusieurs moustiques ou d’un seul moustique). Pour se faire, on s’appuie sur la loi de Bernoulli avec une probabilité p = MAF pour chaque génotype (0/1). Zéro (0) est considéré comme un succès et 1 échec dans cette situation. Les génotypes « 0/0 » sont directement recodés par 0 et «1/1 » par 1.
Cependant, nous avons simulé ces données 100 fois sur cette base, en utilisant le fichier contenant les données manquantes. Et à la fin de chaque simulation, nous avons calculé la corrélation entre la fréquence de l’allèle mineur avant et après l’imputation et le phasing pour chaque bSNPs afin de conserver le fichier avec la corrélation la plus élevée.
Structure des populations
Une fois les données recodées, nous avons étudié la structure de la population, c’est-à-dire de regrouper les différents échantillons en différents groupes qui sont génétiquement similaires.
D’abord, sur les bSNPs, les données manquantes ont été filtrées sur les locus et les échantillons. Le jeu de données final contient uniquement des locus et des échantillons qui ont moins de 20% de données manquantes (figure 11). Ensuite, nous avons fait un clustering de ces données générées avec le package UMAP de python. La figure 12 représente respectivement les différents clusters avant et après imputation des données manquantes et recodage des données hétéro-alléliques. La composition de chaque groupe a été répertoriée dans le tableau s1.
Les deux résultats ont été comparés afin de savoir l’impact de l’imputation et du recodage des données sur l’étude de la structure des populations (figure 13). Voir tableau s3.
Enfin, pour voir si la répartition des échantillons est due à l’effet de données manquantes, nous avons d’abord divisé les échantillons en deux groupes choisis aléatoirement et d’autre part la réduction de la fréquence des données manquantes à 5% puis à 1%. Voir la figure 14.
Diversité génétique des populations
L’étude de la diversité génétique des populations permet de mettre en évidence la différence qui existe entre les différentes sous-populations obtenues après le clustering d’échantillons obtenu après l’étude de la structure des populations. Dans cette étude, nous avons utilisé les données contenant au plus 1% de données manquantes sur les locus et sur les échantillons. D’abord les valeurs FST (indice de fixation) entre les deux sous-populations ont été calculées pour chaque locus avant et après simulation en utilisant la fonction wcFst du package vcflib version 1.0.2 dans le but de savoir s’il y a une grande différentiation entre les deux sous-populations ou pas. Ensuite, nous avons calculé les p-values sur chaque locus avant et après simulation en utilisant la fonction pFst du package vcflib version 1.0.2 dans le but de détecter les SNPs qui créent plus de différenciation entre les deux sous-populations (figure 15). Ces différents SNPs ont été renseigné dans le tableau 4.
Répartition des mSNPs et Indels sur le génome
En génétique ou bien en génomique, un caryogramme est utilisé pour localiser les variantes dans le génome. Étant donné que la distribution des SNPs ou indels sur le génome peut ne pas être uniforme, nous avons réalisé le caryogramme des bSNPs, mSNPs and indels. Nous avons ainsi remarqué que la répartition de ces rares variantes est uniforme sur l’ensemble du génome. Comme le nombre de bSNPs est beaucoup plus important, nous les avons omis pour plus de visibilité entre les mSNPs et les INDELs sur la figure 5A. Sur cette dernière, on observe que les mSNPs sont fortement représentés au début et à la fin de certains chromosomes. Aussi, il y a certaines régions où il n’y a pas de variantes.
C’est le cas au niveau des chromosomes 4, 7, 12 comme illustrés sur la Figure 5B. Et la figure 5C est une illustration de la repartitiion au niveau du chromosome 1.
Les nouvelles variantes
Variation du nombre de nouvelles variantes par rapport à 1984
En 1984, la chloroquine fut introduite pour traiter les cas de paludisme dans beaucoup de pays du monde et notamment en Afrique sub-saharienne. Cette molécule a permis d’avoir beaucoup de succès dans la lutte contre la malaria. Cependant, le parasite a su développé un mécanisme interne qui lui a donné une capacité à résister contre la chloroquine. C’est le début de l’intensification des recherches pour identifier les mutations responsables de cette résistance. En considérant 1984 comme année de référence, nous avons déterminé l’ensemble des nouvelles variantes comparées à chacune des autres années de collecte. Les résultats obtenus attestent qu’il n’y a pas de nouvelles variantes en 1990 et en 2000 comparé à 1984. Pour les autres années, le nombre de nouveaux SNPs et INDELs compare à 1984 est plus élevé en 2008, 2014 et 2015 (figure 7A). Pour chacune des années comparées à 1984, le pourcentage de nouvelles bSNPs reste presque deux fois plus grand que celui des mSNPs et indels (figure 7B).
Pour chaque SNPs ou INDELs, nous avons calculé la fréquence de l’allèle alternatif dans le but de suivre leur évolution au cours du temps. Même s’ils sont moins nombreux que les bSNPs, la fréquence des INDELs et les mSNPs est majoritairement plus élevée (figure 7C, 7D et 7E).