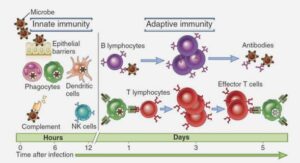
Étude de la variabilité de réponse immunitaire innée chez l’Homme
Les forces démographiques
La dérive génétique
En l’absence de sélection naturelle et de migration, les fréquences alléliques au sein d’une population varient de manière aléatoire (Nei, 1987; Wright, 1931). Ce phénomène est appelé dérive génétique. Ses effets sont d’autant plus importants que la taille de la population est restreinte. En effet, l’ensemble des allèles présents dans une population à une génération donnée n’est que le fruit d’un échantillonnage aléatoire du réservoir allélique de la génération précédente. Pour que la totalité des allèles d’une génération soit transmis à la suivante, il faut que le nombre de descendants tende vers l’infini. Les fréquences alléliques restent dans ce cas stables au cours des générations. Plus le nombre d’individus pouvant se reproduire dans une population où les rencontres sont aléatoires, appelé taille efficace de la population ou Ne, est faible, plus on se s’éloigne de cette situation. L’échantillonnage se fait alors dans un réservoir très restreint, ce qui peut aboutir à d’importantes différences de fréquences alléliques entre deux générations. Ainsi, en suivant ce modèle, une mutation qui vient d’apparaître peut : (i) être éliminée, (ii) augmenter en fréquence jusqu’à fixation (sa fréquence aura atteint 1) ou (iii) être maintenue dans la population au cours du temps. Les effets de la dérive génétique sont particulièrement importants lors de l’apparition d’une espèce, après un goulot d’étranglement, ou dans le cadre d’un isolat génétique. Dans l’ensemble de ces conditions, la diversité génétique sera réduite (1000 Genomes Project Consortium, 2010; Peltonen et al., 1995).
Les migrations
La migration correspond au déplacement d’un groupe d’individus de son lieu de vie vers une nouvelle zone géographique. Ce groupe peut créer une nouvelle population ou se mélanger avec une population déjà installée à cet endroit. Contrairement à la dérive génétique, les migrations n’affectent pas les fréquences alléliques au niveau de l’espèce entière. En revanche, elles peuvent les modifier 11 indirectement dans une population donnée. Par exemple, les migrations peuvent mener à une réduction de la taille efficace de la population dans laquelle les effets de la dérive génétique seront importants. À l’inverse, si le déplacement d’individus est suivi d’un mélange avec une population préexistante, la diversité génétique de la population receveuse va augmenter. On parle alors de flux génétique ou d’introgression (voir section 1.2.2.3). Ces dernières années, de nombreuses études ont porté sur la diversité génétique présente sur l’ADN mitochondrial ou le chromosome Y dans le but de reconstruire l’histoire migratoire de notre espèce (Underhill and Kivisild, 2007). Les marqueurs génétiques portés par ces séquences d’ADN n’étant présents qu’en une seule copie dans les cellules de chaque individu, ils s’affranchissent des évènements de recombinaison méiotiques et ne sont soumis qu’à l’apparition de nouvelles mutations. En tenant compte du taux de mutation, il est donc possible de reconstruire la généalogie des haplotypes retrouvés dans le génome mitochondrial et sur le chromosome Y et de dater les nœuds des arbres phylogénétiques ainsi obtenus (Cann et al., 1987; Cavalli-Sforza and Feldman, 2003; Underhill and Kivisild, 2007). Les données obtenues grâce à cette stratégie montrent que notre espèce est apparue il y a 100 000 – 200 000 ans en Afrique (Cavalli-Sforza and Feldman, 2003; Chen et al., 1995; Ingman et al., 2000; Ingman and Gyllensten, 2001; Underhill and Kivisild, 2007). Elles soutiennent le modèle de « remplacement rapide » qui stipule que les populations non africaines modernes résultent de la sortie côtière d’un ancêtre commun d’Afrique subsaharienne vers l’Asie et le reste du monde (Figure 2, (Cann et al., 1987; Cavalli-Sforza and Feldman, 2003; Gunnarsdóttir et al., 2011; Jobling and Tyler-Smith, 2003; Macaulay et al., 2005; Quintana-Murci et al., 1999; Thangaraj et al., 2005; Underhill and Kivisild, 2007). Des données autosomales obtenues récemment (Excoffier et al., 2013; Fagundes et al., 2007; Gravel et al., 2011; Hellenthal et al., 2008; Laval et al., 2010) mettent en évidence la grande diversité génétique observée dans les populations africaines par rapport aux autres populations ainsi que le fait que la diversité présente hors Afrique semble n’être qu’un échantillonnage de celle observée dans ce continent (1000 Genomes Project Consortium, 2010; International HapMap 3 Consortium, 2010; International HapMap Consortium, 2007), corroborant aussi ce modèle.
Flux génétique des hommes archaïques à l’homme moderne
Comme nous l’avons mentionné précédemment, les migrations ont joué un rôle majeur dans l’histoire évolutive de l’espèce humaine. Le modèle de la sortie d’Afrique aujourd’hui communément admis suggère qu’au cours de sa colonisation de l’Eurasie et de l’Océanie, l’Homme moderne s’est retrouvé au contact de groupes archaïques qu’il a peu à peu supplantés. Des données archéologiques montrent que l’Homme de Néandertal était présent en Europe et en Asie de l’Ouest il y a 40 000 ans (Higham et al., 2014). La comparaison de cette estimation avec la datation des sites archéologiques associés aux Hommes modernes les plus anciens en Europe suggère qu’Homo sapiens et Homo neanderthalensis ont pu cohabiter pendant 2 600 ans au moins dans cette région (Higham et al., 2014). Ces dernières années, les techniques de séquençage d’ADN anciens ont été grandement améliorées et les séquences de l’intégralité des génomes de plusieurs représentants de groupes d’Hommes archaïques sont aujourd’hui disponibles (Castellano et al., 2014; Green et al., 2010; Meyer et al., 2012; Prüfer et al., 2014; Reich et al., 2010). Ces données ont permis d’établir que des croisements entre Hommes modernes non africains et Hommes archaïques se sont faits il y a 50 000 à 60 000 ans (Fu et al., 2014; Sankararaman et al., 2012; Seguin-Orlando et al., 2014). Les Hommes de Néandertal ont ainsi contribué au Figure 2. Origines de l’Homme moderne et routes migratoires déterminées à partir de données génétiques. 13 génome des populations eurasiennes à hauteur maximale de 4% et les Dénisoviens ont contribué au génome des populations malaisiennes modernes à un taux de 6% (Green et al., 2010; Prüfer et al., 2014; Reich et al., 2010). Le fait que les populations asiatiques présentent une introgression d’allèles archaïques plus importante atteste d’une histoire démographique complexe qui pourrait impliquer un second évènement d’introgression dans ces populations ou d’une dilution des allèles de Néandertal dans les populations européennes due à un mélange avec une population ancestrale non identifiée (Vernot and Akey, 2015). Une étude récente atteste aussi d’un faible taux d’introgression d’allèles Dénisoviens dans les populations natives américaines et celles d’Europe de l’Est (Qin and Stoneking, 2015). Enfin, de récents travaux suggèrent que certaines populations africaines portent elles aussi des traces de mélanges génétiques avec des groupes d’Hommes archaïques encore indéterminés (Hammer et al., 2011; Lachance et al., 2012; Plagnol and Wall, 2006). Différentes méthodes permettant d’estimer l’intensité du flux de gènes archaïques dans le génome des populations modernes ont été développées ces dernières années (pour une revue, voir Racimo et al., 2015). L’utilisation d’une de ces méthodes, dont les principes sont détaillés en Figure 3, a révélé que l’introgression n’est pas homogène le long des génomes des populations européennes, asiatiques et océaniennes. En effet, la majorité des régions codantes présentent un faible taux d’introgression archaïque (Sankararaman et al., 2014). Cette structure est aussi observée dans les gènes fortement exprimés dans les testicules et le chromosome X ne porte que peu d’allèles introgressés. Ces données suggèrent que les allèles provenant des Hommes archaïques ont été en partie purgés chez les Hommes modernes à cause de la diminution de la fertilité des descendants mâles issus de croisements entre Homo sapiens et Hommes de Néandertal (Sankararaman et al., 2014). À l’inverse, des haplotypes introgressés dans des séquences codantes ont été maintenus chez les Hommes modernes. Par exemple, certains allèles provenant de Néandertal retrouvés dans les génomes des populations non africaines sont liés à des fonctions immunitaires (Sankararaman et al., 2014). Des haplotypes archaïques couvrant des gènes tels que HLA, OAS et STAT2 sont trouvés à fortes fréquences dans les populations européennes, asiatiques et malaisiennes (Abi-Rached et al., 2011; Ding et al., 2014b; Mendez et al., 2012a, 2012b; Mendez et al., 2013; Temme et al., 2014), même si ces données sont parfois sujettes à controverse (Ding et al., 2014a).
Table des figures |