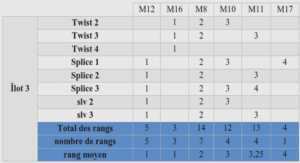
Complémentarité enzyme
La carboxypeptidase est une enzyme digestive qui hydrolyse la liaison peptidique en position C-terminale. L’hydrolyse n’intervient que si l’acide aminé présente une chaîne latérale aromatique ou une chaîne aliphatique volumineuse. Cette enzyme comporte une seule chaîne de 307 acides aminés. Sa structure est globuleuse et sa forme est globalement celle d’une ellipsoïde (38% d’hélices alpha et 17% de feuillets bêta). La zone de reconnaissance est formée d’une large poche située près de la surface. Elle est bordée par trois acides aminés (arginine 145, tyrosine 248, acide glutamique 270). En présence du substrat, la zone de reconnaissance subit des modifications spatiales importantes : les chaînes latérales des trois acides aminés de rapprochent du substrat, en particulier la chaîne latérale de la tyrosine 248 se rabat tel un clapet. La conséquence de ce déplacement (évalué à 1,2 nm) est la fermeture du site catalytique et l’expulsion de molécules d’eau, point de départ de l’hydrolyse du substrat. La zone catalytique est située dans un sillon, à proximité immédiate de la zone de reconnaissance. Elle est formée d’un ensemble tétraédrique constitué de deux histidines (His 196 et His 69), d’un acide glutamique (Glu 72) et d’une molécule d’eau. Au centre de ce tétraèdre, un ion zinc (Zn2+) constitue l’élément essentiel de l’activité de l’enzyme.
La carboxypeptidase (cpa) est une enzyme digestive qui hydrolyse certaines liaisons peptidiques (entre deux acides aminés d’une protéine). Le fichier cpa.pdb représente la carboxypeptidase isolée. Le fichier cpa-sub.pdb représente la carboxypeptidase associée à son substrat (sub). Ce dernier est ici représenté par un dipeptide Glycine-Tyrosine (Gly1, Tyr2).Clic gauche : rotation de la molécule (glisser). Clic droit : translation de la molécule (glisser). Maj + clic gauche : zoom avant ou arrière (glisser). Maj + clic droit : rotation de la molécule dans le plan (glisser). On peut aussi faire tourner la molécule dans chacune des trois directions de l’espace en utilisant les curseurs situés en bas à gauche de l’écran. Les icônes permettent de choisir le mode de représentation de la molécule active. Quand une molécule est complexe vous avez intérêt à choisir au départ une représentation simple comme Rubans. Cela ne vous empêche pas de changer de représentation par la suite (Sphères, Bâtonnets…). Une modification de représentation ne s’applique qu’à la zone sélectionnée d’une molécule. Quand on ouvre une molécule elle est entièrement sélectionnée. L’icône (Afficher la sélection) permet de vérifier les zones sélectionnées. Les icônes permettent de modifier globalement la sélection.
Les icônes permettent de ne sélectionner qu’une partie de la molécule. Après en avoir activé une Cliquer sur la zone à sélectionner puis changer la représentation et/ou colorer. – Soit cliquer sur (Palette). Dans la fenêtre Colorer choisir successivement ce que vous voulez colorer et une couleur. – Soit, dans le menu Atomes, choisir Colorer par (Shapely (acide aminé), Chain (Chaîne) …). Dans le menu Liaisons choisir Liaisons hydrogène / Afficher et/ou Ponts disulfure / Afficher. Le Type / Bâtonnets de Rayon 1 est le plus lisible quand on affiche en rubans. Sélectionner toute le molécule en cliquant (Sélectionner tout) puis (Bâtonnets). Dans le menu Editer choisir Commande et écrire Restrict within(6.0,Gly1) ou bien Restrict within(6.0,Tyr2). Gly1 et Tyr2 sont les acides aminés du substrat et 6.0 représente un rayon de 0,6 nm. – Soit positionner le curseur sur un acide aminé et lire son nom et sa position en bas de l’écran (légende, voir document 2). – Soit cliquer sur (Label) puis sur le ou les acides aminés (légende, voir document 2). Effacer éventuellement les labels (menu : Atomes / Labels / Effacer) Sélectionner toute le molécule en cliquant (Sélectionner tout) puis (Bâtonnets). Pour chacune des deux molécules, dans le menu Editer choisir Commande et écrire Restrict 69,72,145,196,248,270 (sans espaces après les virgules) puis cliquer OK. Pour cpa-sub.pdb faire ensuite apparaître le substrat Cliquer sur (Expression), écrire dans la fenêtre : *s, puis cliquer OK et (Fil de fer). On peut aussi taper Select *s dans la fenêtre Editer / Commande. L’imprimante de la salle de TP est en noir et blanc. Grâce à l’icône (Palette) ouvrir la fenêtre Colorer puis choisir Fond / Blanc puis imprimer. ATTENTION. Une impression en fond noir est une erreur de manipulation. Possibilité d’imprimer deux images sur la même feuille en passant par le logiciel WORD.