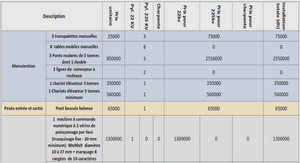
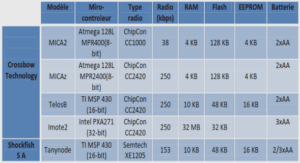
Pourquoi reconstruire des génomes ancestraux ? D’un point de vue fondamental, les études des systèmes biologiques contemporains en utilisant, par exemple, des approches d’anatomie, de biochimie, de physiologie, et biologie moléculaire, sont sérieusement limitées par l’absence d’un cahier de laboratoire de l’Évolution qui décrirait et expliquerait leur mise en place, leur organisation et leur fonctionnement. L’objectif à long terme de posséder les génomes ancestraux est d’établir un vaste cadre d’étude de l’évolution pour corriger le manque cruel de données historiques (la molécule d’ADN ne se conserve guère plus d’une centaine de milliers d’années). Pour atteindre cet objectif, de nombreux développements algorithmiques sont nécessaires pour traiter de manière efficace et systématique les larges volumes de données disponibles, selon une méthodologie rigoureuse.
Les données génomiques suivent en général bien ce paradigme parce qu’elles ont une résolution très élevée (à la base près), sont très fiables (beaucoup de séquences de génomes contiennent moins d’une erreur toutes les 10000 bases), très abondantes (plus de 100 génomes eucaryotes séquencés, plus de 1000 procaryotes), et centralisées dans des bases de données publiques. Le génome fournit aussi des points d’entrées fondamentaux vers les propriétés fonctionnelles des organismes, comme la présence ou l’absence de gènes, l’expansion ou la régression des familles de gènes, la topologie des éléments cis-régulateurs, qui en retour nous informent sur la vraisemblance de certaines voies métaboliques ou de développement qui peuvent exister dans un organisme, et l’importance des fonctions spécifiques à chaque espèce. Les génomes représentent ainsi la fondation sur laquelle de nombreuses avancées peuvent être faites, et accéder à ces informations dans un génome ancestral fournit un large spectre de ces propriétés.
D’un point de vue plus pratique, compte tenu de la quantité astronomique de données génomiques apportées à la communauté, dont le rythme, vraisemblablement, s’accélèrera encore dans les prochaines années, il est critique de garder un degré d’organisation substantiel pour la distribution et la présentation des données. Les résultats de reconstructions de génomes ancestraux permettront de lier naturellement les séquences et les annotations des espèces modernes entre elles, et avec celles des espèces ancestrales, dans le sens de l’évolution, en suivant la phylogénie des espèces. Les génomes ancestraux serviront de points de référence unique pour comparer des génomes descendants, ce qui facilitera grandement l’identification de propriétés génomiques ancestrales, et donc les gains ou pertes lignées-spécifiques. Réciproquement, les résultats qui continueront à être obtenus avec les différents organismes modèles les enrichiront en retour. Pour résumer, les génomes ancestraux représentent les fondations qui nous aideront à déchiffrer les différentes composantes moléculaires contribuant à l’évolution des espèces, et qui ont mené à une telle variété d’espèces et de systèmes biologiques .
Les premières expériences de génomique comparative datent de plus de 30 ans, et ont ouvert la possibilité de comparer les chromosomes (morphologie et bandes chromosomiques) à partir de cellules en métaphase. Ces techniques ont fourni les premières données pour permettre de déterminer les réarrangements ancestraux de chromosomes chez les vertébrés [Rumpler et Dutrillaux, 1976, Yunis et Prakash, 1982]. Plus récemment, des expériences d’hybridation fluorescente in-situ (FISH : fluorescence in situ hybridisation) entre espèces ont été développées et ont permis d’améliorer fortement la précision et la portée de ces approches. Dans une expérience typique, l’ADN d’un chromosome donné d’une espèce de référence (souvent l’homme) est purifié, marqué par fluorescence, découpé, puis hybridé sur tous les chromosomes d’une autre espèce, cible. Il est même possible d’étudier la répartition de plusieurs chromosomes de l’espèce référence à la fois en utilisant plusieurs types de marqueurs fluorescents. Cette technique s’appelle Chromosome painting ou encore Zoo-FISH [Scherthan et al., 1994, Wienberg et al., 1990]. L’analyse des images au microscope permet de découvrir des différences chromosomiques de grande échelle (de l’ordre du mégabase), comme les fusions, les fissions, ou les translocations d’une grande région. En revanche, les translocations de petites régions ne sont pas détectables, et les inversions (intra chromosomique) sont, par principe de l’expérience, impossibles à repérer.
Classiquement, les expériences révèlent le nombre de segments chromosomiques de l’espèce cible qui correspondent à un chromosome (entier) de l’espèce de référence. Si ce nombre est 1, alors l’ancêtre commun de ces deux espèces devait certainement posséder ce chromosome d’un seul tenant (Figure 2.1.A). En revanche, si ce nombre est 2 ou plus (Figure 2.1.B), alors, il est nécessaire d’utiliser les données de comparaisons avec d’autres espèces (éventuellement des outgroups) pour décider quel état est ancestral (la version contiguë de l’espèce de référence ou la version fragmentée de l’espèce cible). Ensuite (Figure 2.2), on peut étudier des associations de chromosomes de l’espèce de référence dans l’espèce cible. Selon le même principe, si deux chromosomes de l’espèce de référence s’hybrident sur des chromosomes différents, alors ils devaient certainement être sur des chromosomes différents chez l’ancêtre. Si, au contraire, ils s’hybrident sur le même chromosome de l’espèce cible , alors, il est nécessaire d’utiliser d’autres données de comparaisons pour décider de l’état ancestral et de la position temporelle d’un réarrangement de translocation (voire une fusion ou une fission).
Le raisonnement sous-jacent pour déduire les réarrangements et l’état ancestral suit en général les principes de la cladistique [Dobigny et al., 2004], et les décisions prises pour définir un caractère comme ancestral sont basées sur la parcimonie. Dans la plupart des cas, les données sont analysées à la main (les nombres d’espèces et de réarrangements impliqués sont généralement limités), mais certaines situations, rares, avec des données en grande quantité ou complexes, demandent une analyse informatique : le logiciel PAUP est alors utilisé [Müller et al., 2003].
Le Zoo-FISH est extrêmement puissant car virtuellement, n’importe quelle espèce peut être étudiée, sans demander de technologie particulière (tel le séquençage) : la seule ressource nécessaire est un échantillon de tissu à partir duquel on peut faire évoluer des lignées cellulaires. Ces analyses ont cependant une limite physico-chimique due à la capacité des molécules d’ADN à s’hybrider. Deux espèces trop éloignées phylogénétiquement ont en général une trop grande divergence moléculaire de leurs chromosomes (mutations, insertions, délétions) et ne peuvent être comparées par cette technique. Ainsi, chez les mammifères, il est possible de comparer les euthériens (mammifères placentaires) entre eux (environ 100 millions d’années de divergence), mais difficilement les euthériens aux métathériens (marsupiaux), sauf pour le cas, unique, du chromosome X [Glas et al., 1999, Wienberg, 2004].
La première reconstruction d’un caryotype ancestral en utilisant des données cytogénétiques était fondée sur du Zoo-FISH entre l’homme et sept espèces de mammifères [Chowdhary et al., 1998]. Depuis lors, les résultats de nombreuses études ont permis de retrouver l’organisation ancestrale des chromosomes dans différents ancêtres, en s’attachant particulièrement aux quatre clades de mammifères placentaires [Richard et al., 2003, Yang et al., 2003, Froenicke, 2005, Yang et al., 2006, Ferguson-Smith et Trifonov, 2007, Stanyon et al., 2008, Westerman et al., 2010]. La base de données ChromHome [Nagarajan et al., 2008] recense une partie des résultats des comparaisons cytogénétiques entre espèces de mammifères. On peut toutefois rappeler la possibilité d’études cytogénétiques en dehors des mammifères, comme Schneider et al. [2009] chez des espèces de scorpions, à condition que les génomes n’aient pas encore trop divergé.
Le E-Painting [Kemkemer et al., 2006, 2009] est une méthode hybride entre les manipulations de cytogénétique et les données issues du séquençage des génomes. Les marqueurs conservés (typiquement des paires de gènes orthologues) permettent de simuler l’hybridation de sondes, et ainsi de comparer deux génomes. Il est donc possible, en appliquant les mêmes principes, d’atteindre une résolution jamais atteinte jusqu’alors. Cependant, le risque d’erreur est fortement accru car le nombre d’espèces séquencées (et donc comparées) n’est pas aussi important que pour la cytogénétique «classique». Les branches espèce-spécifiques sont donc beaucoup plus longues, ce qui augmente la chance que les mêmes réarrangements aient eu lieu sur des lignées indépendantes (homoplasie), et les erreurs de reconstructions. Malgré ceci, les reconstructions les plus récentes [Kemkemer et al., 2009] concordent nettement avec les prédictions de la cytogénétique.
I Introduction |